135-一次理解一张图—coefficients of variation
刘小泽写于19.8.24 因为经常看的下面👇这样的图,因此有必要看看它到底代表什么意思,有什么用? 另外我发现,**看懂一个图,关键有两个:**一个是对图中点的认识度有多少:是代表基因还是样本;另一个是横纵坐标,为什么要这么设定(比如使用了log),以及其中的公式(例如mean、CV、sd等)
图片来自:https://jdblischak.github.io/singleCellSeq/analysis/cv-adjusted.html
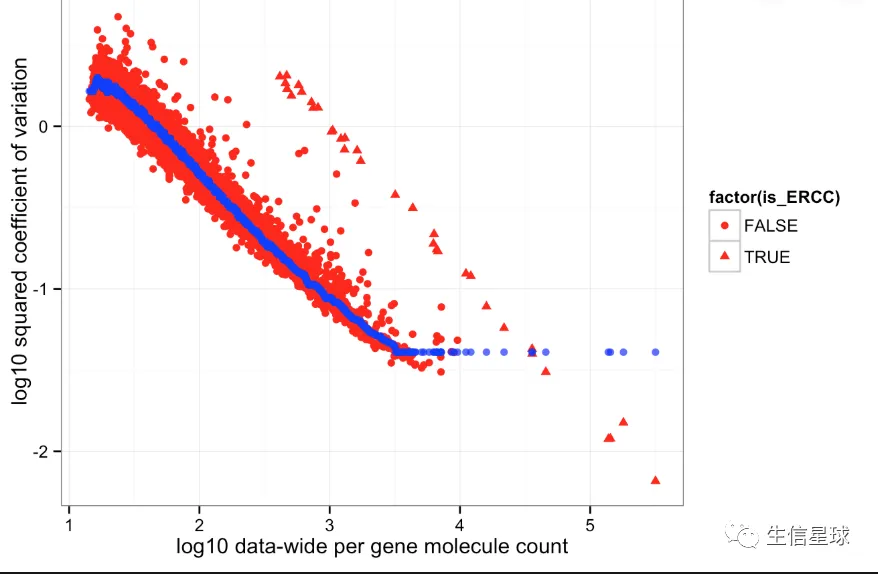
(剧透:上图中的纵坐标意思就是:基于count计算的方差与均值的比值再取log10,继续往下看)
简单理解
下面来自:https://www.investopedia.com/terms/c/coefficientofvariation.asp
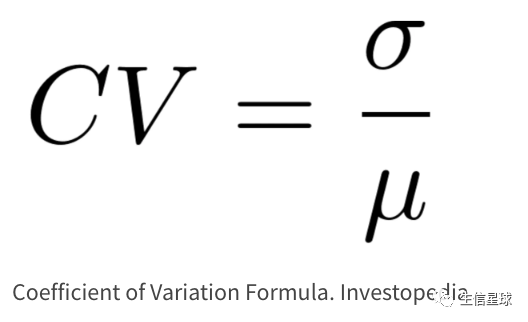
Coefficient of variation (CV)中文翻译是变异系数,它是标准差与均值的比值
标准差就是每个观测值减去均值,得到一系列的差值,再求这些差值的平方和,最后再开方就得到了标准差
这个值越大,均值附近的数据分布越分散;值越小,约接近观测值
如果两组数据之间数据均值差别很大(比如一个是10,一个是10000),也就是说它们的数据是不同维度的,不能直接比较,但是用了CV值就相当于它们各自对自己数据进行了转换,落在相似的范围中,就能进行比较
如果数据的均值接近0,那么CV基本就没办法反映真实分布,这样会导致CV趋近于正无穷
下面来自:https://influentialpoints.com/Training/Coefficient_of_variation_Use_and_misuse.htm
- 如果两组数据的观测值在一个数量级,那么可以用标准差来比较它们的离散度
- 如果两个数据差别太大(比如老鼠和大象的重量),然后就要用CV
- 可以在一张图中比较不同部分的离散度(就像我们这里的第一张图,它就比较了ERCC和内源基因的离散度,其中每个点都是一个基因。ERCC衡量了技术误差(比如PCR扩增偏好),如果我们自己的内源基因比ERCC分布还稳定,说明内源基因的技术误差很小
下面来自一篇文章:https://bmcgenomics.biomedcentral.com/articles/10.1186/s12864-015-1806-8
它介绍了使用低丰度转录本,对比barcode和原始的reads count来说明barcode的好处
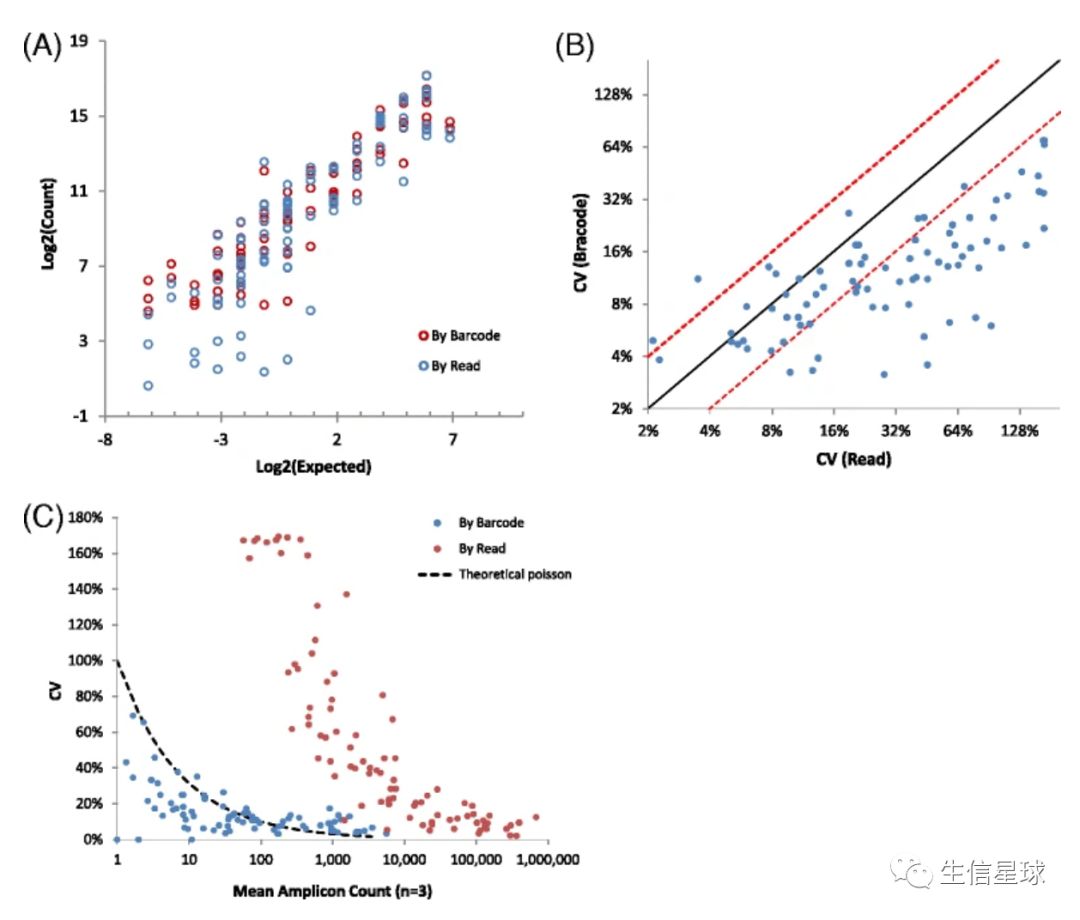
- 每个点表示一个扩增子
- 其中图a能看出两件事:一个是越往右上方(说明得到的转录本越多),barcode和raw reads得到的转录本更接近;另一个是当转录本比较少时(图中左下方),barcode得到的转录本和raw reads得到的转录本数量相关性下降,并且看到barcode比raw得到的转录本更多。因此文章建议:对于低丰度转录本定量,使用barcode更加可信
- 其中图b看到:Read的CV比barcode的CV总体更大,说明使用Read得到的转录本的技术误差更大
- 图c看到:蓝色的点(barcode count)整体在红色点(read count)下方,并且绘制了泊松分布曲线(理论上,短序列测序得到的覆盖深度符合泊松分布),看到barcode的CV与理论值更接近
再回到第一张图
看图例,三角形的点是ERCC,圆形的点是内源基因,一般ERCC是表达量很高并且很稳定的,可以用来指示技术误差(就是说,如果已经加入了稳定外源的RNA,表达量结果依然波动很大,就说明不是受到生物因素的干扰,而是外在操作的技术问题,比如PCR扩增偏好)。
使用CV统计指标比较ERCC和内源基因,也是为了兼顾高、低两种维度的数据(ERCC表达量一般都成百上千,而内源少的只有几个。如果是单细胞数据,更多的会是0)
然后我们看到我们的内源基因CV分布比ERCC的CV分布更偏下,也就是比ERCC分布更稳定
还有一张类似的:
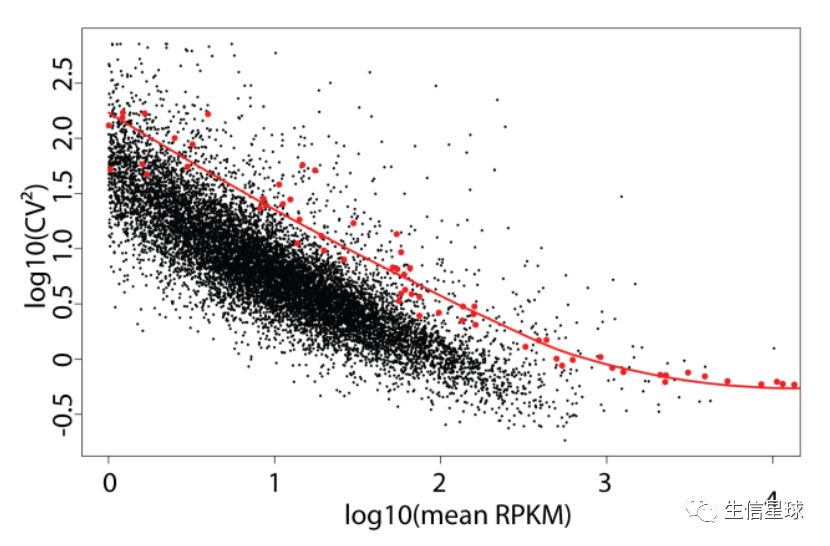
CV-mean图主要还是为了质控,至于怎么做出来的,可以看这篇:https://www.jianshu.com/p/3525e624946a