197-也许是最方便的SRA数据下载方式
刘小泽写于2020.6.15
之前下载数据会自己去GEO找sra-selector,然后下载SRA后使用fastq-dump转为fq,或者可以自己去找fastq的下载链接,直接下载,省去转换数据这一步。但还有更”懒“的方法:自己只需要输入编号,就能获得下载脚本,并且还提供了重命名服务。
今天要介绍的就是一个网页工具:https://sra-explorer.info/#
前言
它是瑞典斯德哥尔摩的Phil Ewels开发的,他的个人主页在:http://phil.ewels.co.uk/
他参与开发的项目包括:MultiQC、ClusterFlow、QC Fail、10XQC等等
SRA Explorer的使用非常简单,也很高效
界面很清爽:
搜索
支持输入:GSE、SRA、SRP、PRJ、EPR,甚至直接通过物种+组织+测序技术搜索
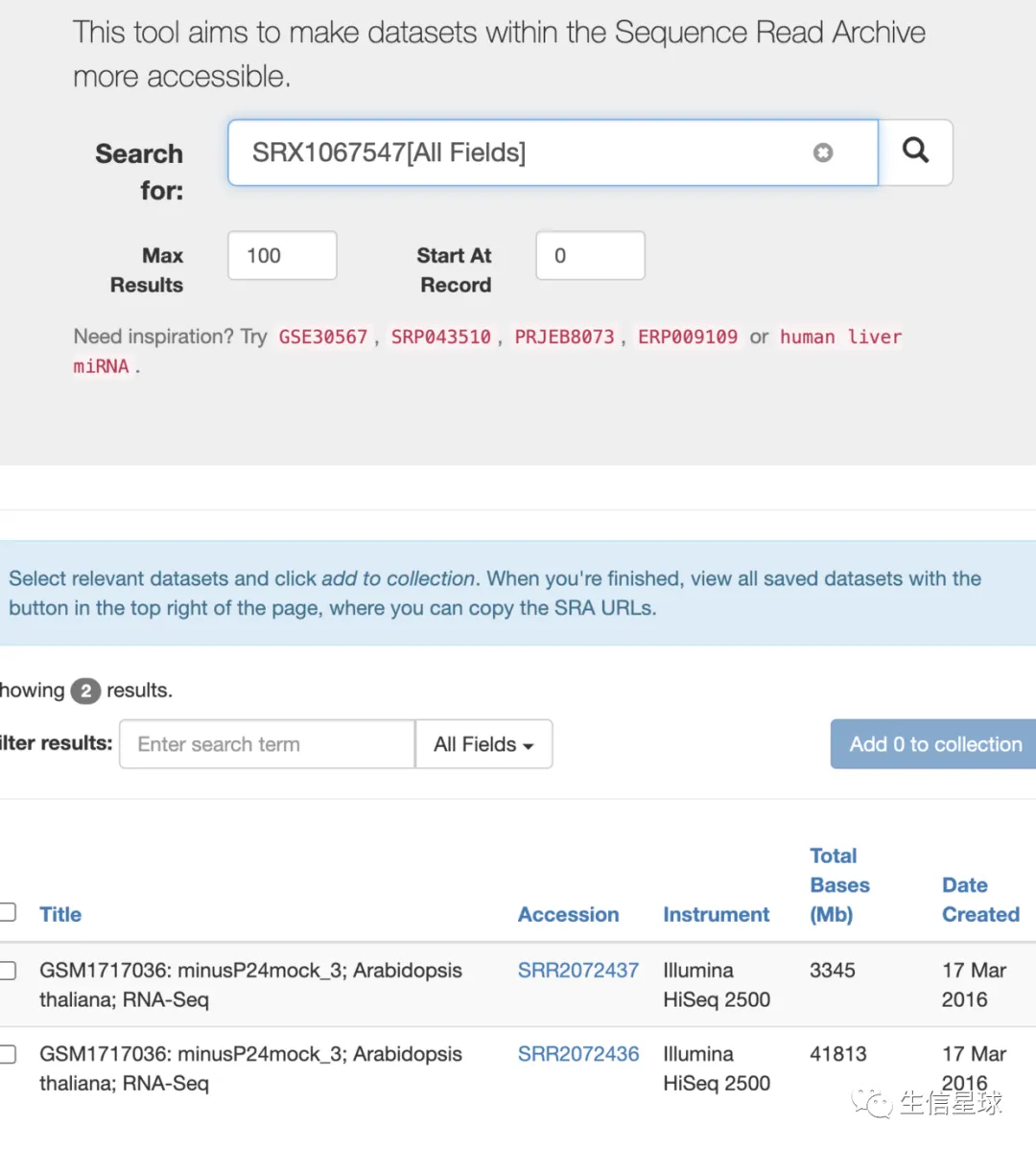
即使一个SRX包括超过一个数据集的时候(多个SRR),也会返回结果,例如:SRX1067547包含两个run
可以组合搜索,但一个词中间不能用空格,比如chipseq不能写成chip seq,否则会被认为是chip + seq组合方式

还可以在一个项目中选取特定测序技术的数据

选好之后,添加到购物车

然后点击上面的:

下载
支持多种下载方式,包括下载fastq、sra,或者下载样本信息
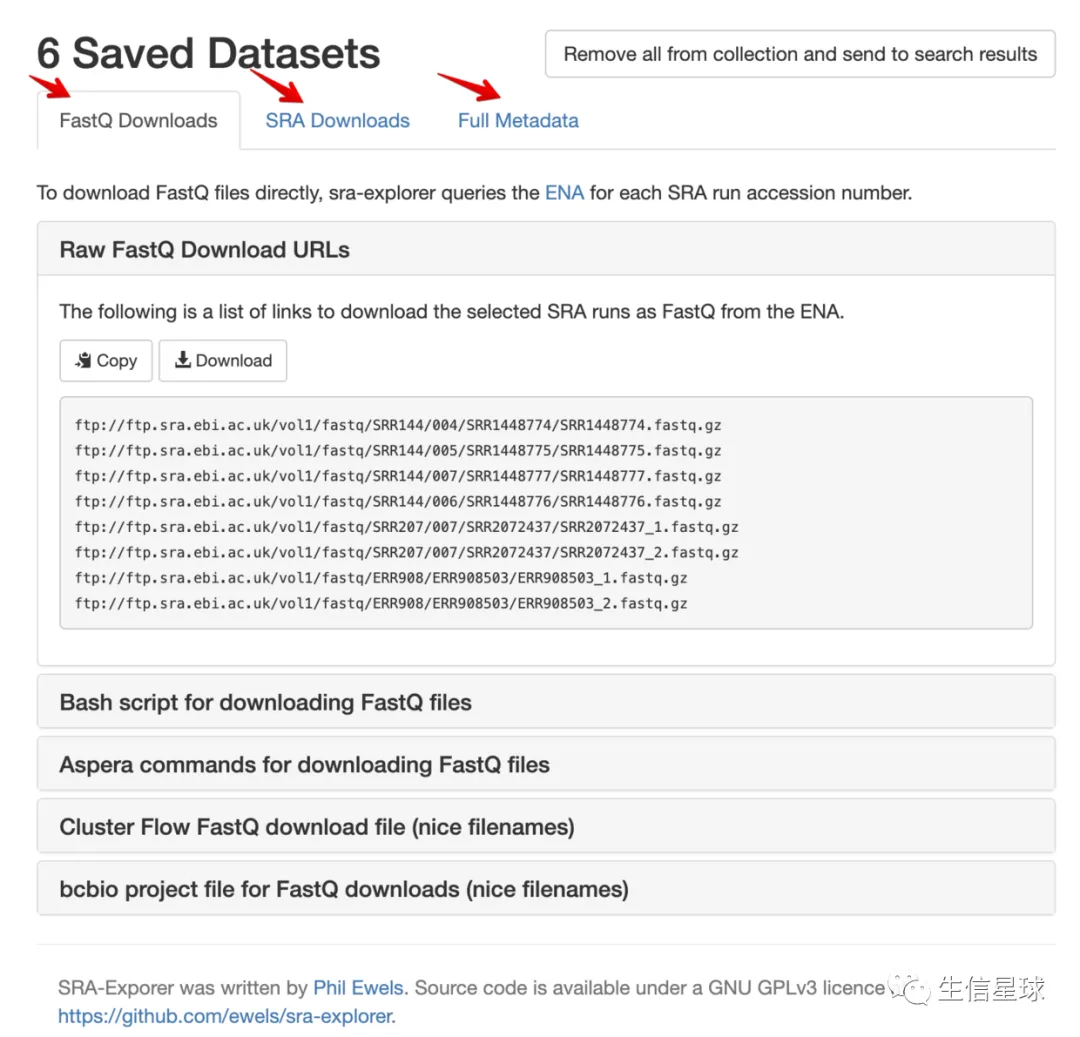
比如下载fastq文件,可以只使用它给出的ftp链接,或者直接下载他的curl -L 普通下载脚本,甚至还贴心给出了Aspera下载链接,还帮忙做了重命名
关于配置aspera可以看: 来吧,加速你的下载
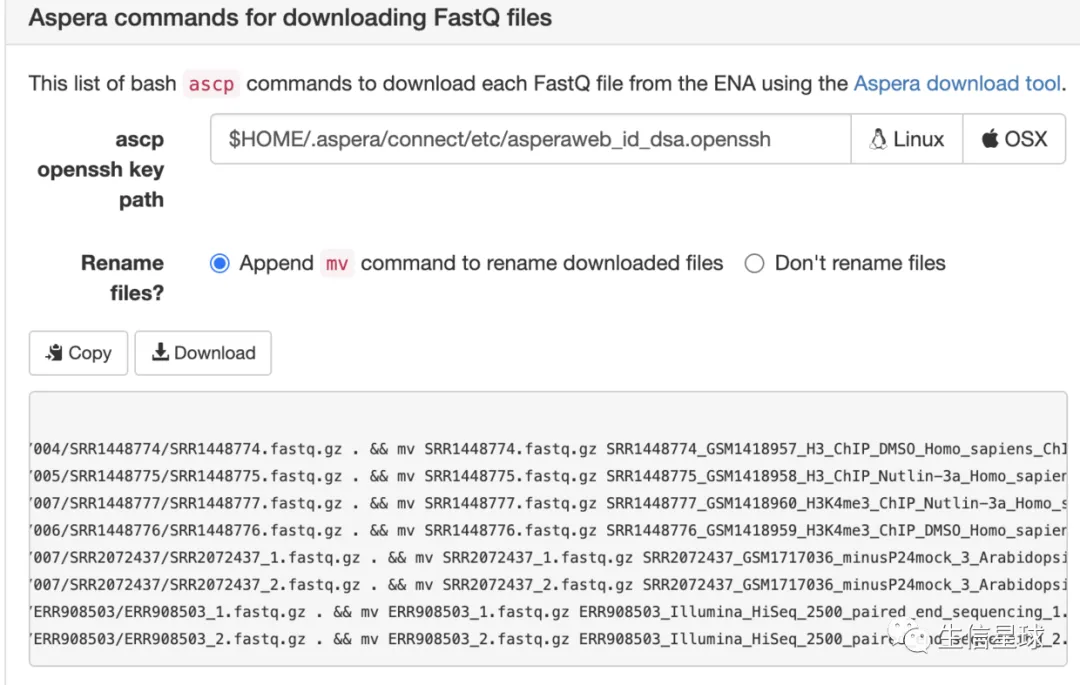
这些虽然都是批量出的脚本,但作者的贴心还是很值得称赞的
后面如果需要样本信息,也可以直接下载到:
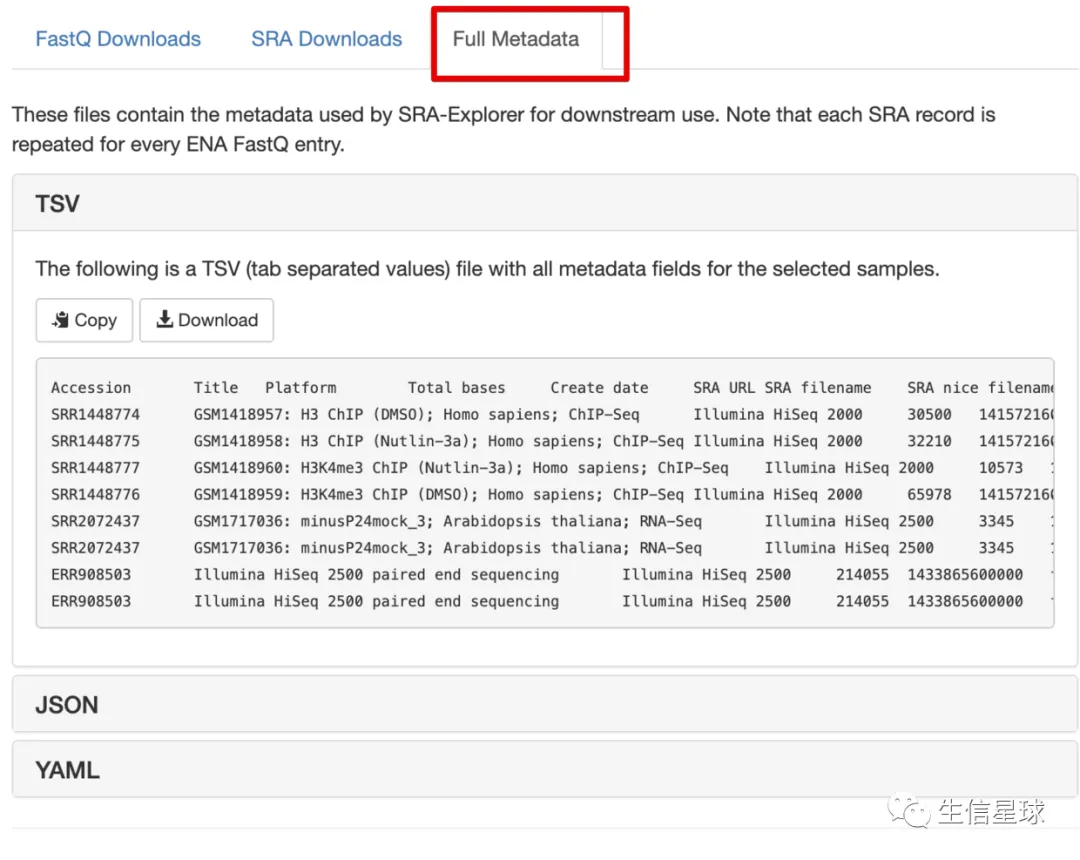
可能存在的问题
如果下载量很大,比如几百个数据,那么要检查一下下载的脚本,会不会遗漏一两个。但是十几个应该还是问题不大的