225-懒惰迫使我学了点API的知识
刘小泽写于2021.1.8 今天晚上绝对称得上是有趣的时光。。。
故事开头
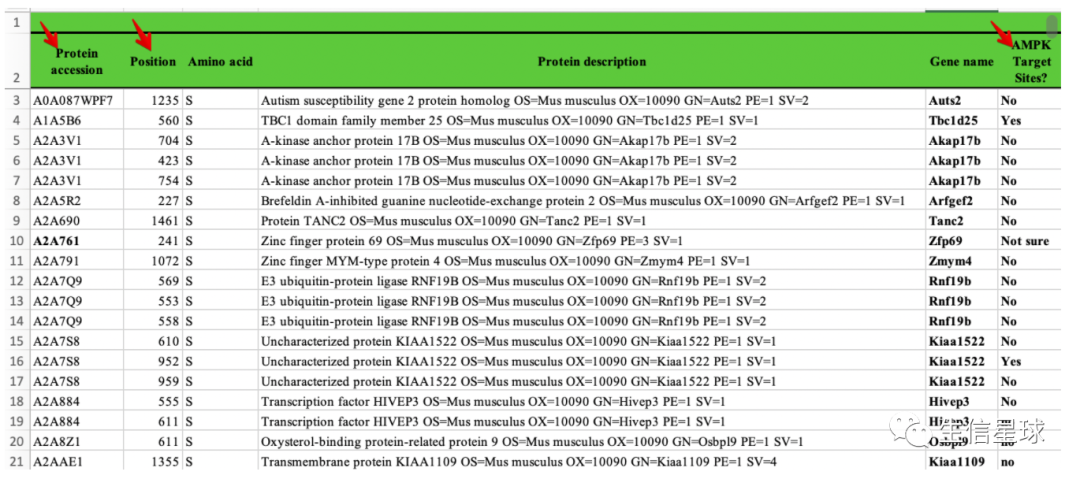
看到这个文件,你可能不知道是啥,我也不知道。有朋友求助我帮他看看第三个箭头处标注的对不对。他说第一个箭头是蛋白ID,第二个箭头是蛋白位点,第三个箭头是该位点是否对应AMP激酶。我猜测这个文件可能是某个实验仪器出来的结果,他是想拿这个结果再与一个网站“Scansite 4.0”进行比较,看看第三个箭头标注“YES”的对不对
开始探索
以第一个标注“yes”的蛋白为例:
A1A5B6,看看网页操作怎么弄
首先打开:https://scansite4.mit.edu/4.0/#home
然后5步走,逐步提交,鼠标点几下,得到结果
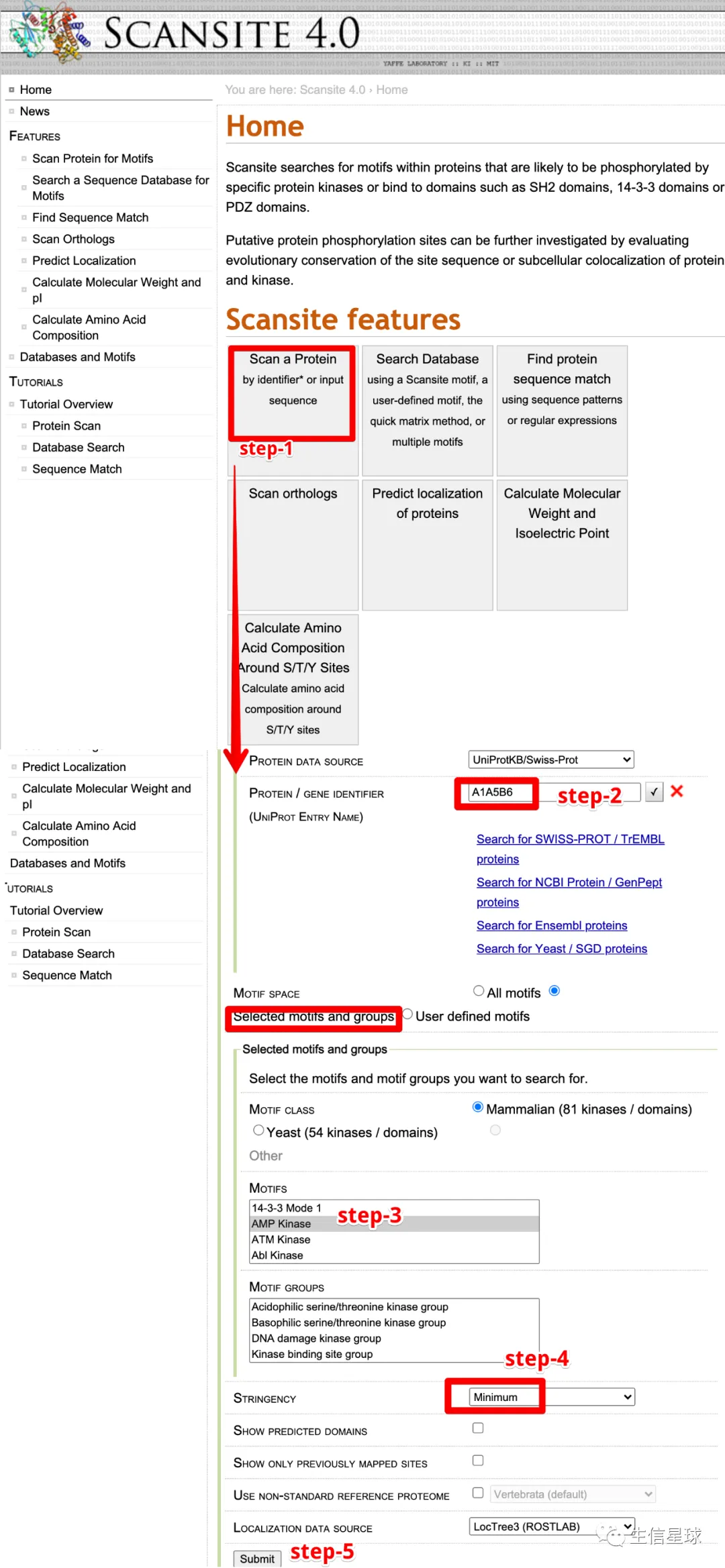
最后看看结果,确实存在560这个位点,于是这个标注“yes”是对的

陷入纠结
上面👆的操作过程,相信大家已经看懂了,很简单,但同时很无趣。
我算了笔账,excel中的”yes“结果有500多个,即使我能稳定快速10s提交一个并且统计完成,也需要500*10/60= 83 min 。这可是我一直不动不吃不喝不上厕所,才能完成的任务
因此,我尝试了3个,就已经陷入”崩溃“的边缘。
我其实想过,这个网站肯定有数据库吧,把相应的数据库下下来,然后放进R就是一马平川。但事实并非如此,找了好半天也没看到有数据提供下载,因为它是在线关联各个数据库的。
求解
偶然翻翻翻,看到了光明:https://scansite4.mit.edu/webservice/


于是,我开始尝试如何利用代码解决问题
首先什么是API?
其中提到了:RESTful ,搜索发现它是API相关的知识。其实API这个名词之前听过,但没怎么用过,也不知道具体是啥。在https://www.smashingmagazine.com/2018/01/understanding-using-rest-api/这里搜索得到:
REST determines how the API looks like. It stands for “Representational State Transfer”. The root-endpoint of Github’s API is https://api.github.com while the root-endpoint Twitter’s API is https://api.twitter.com.
初步感觉,和我们常用的GEO链接替换很相似,比如https://www.ncbi.nlm.nih.gov//geo/query/acc.cgi?acc=GSE52778,替换最后的GSE号就能直达其他的GEO样本
然后仔细阅读网站的帮助文档
大约看了20分钟,我get了网站的xml编写逻辑,按照我的需求,我需要的链接就是:https://scansite4.mit.edu/webservice/proteinscan/identifier=B2RUR8/dsshortname=swissprot/motifclass=MAMMALIAN/motifshortnames=AMPK/stringency=Minimum
链接其中的identifier是可以替换成其他的ID的,这让我有了希望,因为能替换就能循环
打开链接,看到其中确实就是网站显示的结果,重点是我想要site这部分

那么如何获取我想要的site这部分呢?
因此又搜索了:https://www.dataquest.io/blog/r-api-tutorial/
rm(list =ls())
options(stringsAsFactors = F)
# 为避免httr使用过程中掉线,先设置一下
httr::set_config(config(ssl_verifypeer = 0L))
library(httr)
library(jsonlite)
library(stringr)
# 先学会一个
motif='AMPK'
name='A1A5B6'
res = GET(paste0("https://scansite4.mit.edu/webservice/proteinscan/identifier=",name,"/dsshortname=swissprot/motifclass=MAMMALIAN/motifshortnames=",motif,"/stringency=Minimum"))
txt=rawToChar(res$content,)
看下txt,的确很乱,但很有成就感。因为我成功根据我的需求,把网站的内容拿下来了
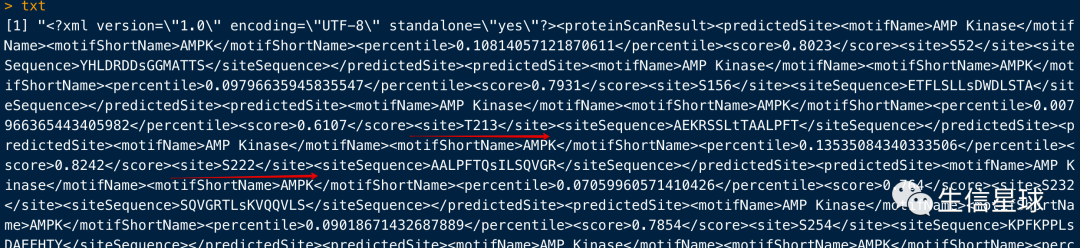
接下来就是,只提取site这部分
result=unlist(str_extract_all(txt, "(?<=<site>).*?(?=</site>)"))
# [1] "S52" "S156" "T213" "S222" "S232" "S254" "S504" "S560" "T586" "S592" "S594" "S609" "S615"
最后,我要循环!
这样,就基本上大功告成了。后续的只是拿网站的结果,与我们自己的结果进行比较即可
result=list()
motif='AMPK'
for (i in 1:length(ac$`Protein accession`)) {
# i=1
name=ac$`Protein accession`[i]
res = GET(paste0("https://scansite4.mit.edu/webservice/proteinscan/identifier=",name,"/dsshortname=swissprot/motifclass=MAMMALIAN/motifshortnames=",motif,"/stringency=Minimum"))
txt=rawToChar(res$content,)
result[[i]]=unlist(str_extract_all(txt, "(?<=<site>).*?(?=</site>)"))
names(result)[i]=name
}
结语
今晚的偶然探索,一是学会了一个网站的使用,更重要的是学习了API在R中的应用。有了这次的代码,之后就不需要手动一个一个输入、查结果、肉眼比对了。试想:如果手动点点点,可能用了一下午才搞定了几百个ID的检查,但人家说能不能把阈值从Minimum设为High再来一遍呢?那时很可能会崩溃。但自从有了代码,不就是重新换个变量名称而已吗?
因此,用代码解决问题,不仅可以提高效率,还能更好地调节心态🤓