074-变异信息那些事
刘小泽写于18.12.31
上篇:主要了解VCF的背景知识; 中篇:VCF怎么来?怎么进行一些小操作来获取内部信息? 下篇:得到变异位点需要做些什么
一般我们会从WES的上游得到SNP、InDel等信息,这些重要的信息都保存在VCF中,那么怎么对这些变异进行提取、评估与解释呢?一起来学习一下
VCF(Variant Call Format)是什么?
之前也写过一篇相关的,这次想要更深层次去了解它https://www.jianshu.com/p/957efb50108f
我们知道,variant calling(找变异)的过程发生在alignment(比对)之后,那么肯定流程更加复杂,因此variant calling得到的结果也要更加精炼、内容更加丰富。于是VCF文件接手了这个棘手的工作。了解VCF,对于想要另辟蹊径发现新研究内容的人来说,真的是一块宝藏,就看你怎么挖掘了。
主要内容
得到一个VCF文件,首先看到的就是它的Header(表头),如下:(其实有非常非常多的头信息…这里只写几行)
##fileformat=VCFv4.1
##FILTER=<ID=LowQual,Description="Low quality">
##FORMAT=<ID=AD,Number=.,Type=Integer,Description="Allelic depths for the ref and alt alleles in the order listed">
##FORMAT=<ID=DP,Number=1,Type=Integer,Description="Approximate read depth (reads with MQ=255 or with bad mates are filtered)">
##GATKCommandLine.HaplotypeCaller=<ID=HaplotypeCaller,Version=3.4-3-gd1ac142,Date="Mon May 18 17:36:4
##INFO=<ID=AN,Number=1,Type=Integer,Description="Total number of alleles in called genotypes">
##contig=<ID=chr1,length=249250621,assembly=b37>
##reference=file:human_genome_b37.fasta
第一行是VCF的版本信息,看似没用,但实际上我们在分析其他数据时,并不能保证一直使用最新的VCF格式,因此检查下VCF版本确保后续提取正确
FILTER行是说过滤了什么内容;
FORMAT和INFO 相当于变异位点的注释信息;
CommandLine 是说使用的call variant工具信息;
contig和reference 是当重复别人数据时,恰好没告诉你数据来源,这时就可以参考这个
接下来才是重点:Records信息
包含至少8列tab分割的常规信息(用来描述变异位点),第9列及以后表示各个样本的变异信息(可以包括成百上千个样本)
前9列信息包括:
CHROM:变异发生的chromosome或者contig
POS:变异发生的基因组坐标(对缺失来讲,显示的是缺失开始的位置)
ID:一般是dbSNP的ID(可有可无)
REF:Forward strand(正链)上的参考等位基因 补充一下基础知识:
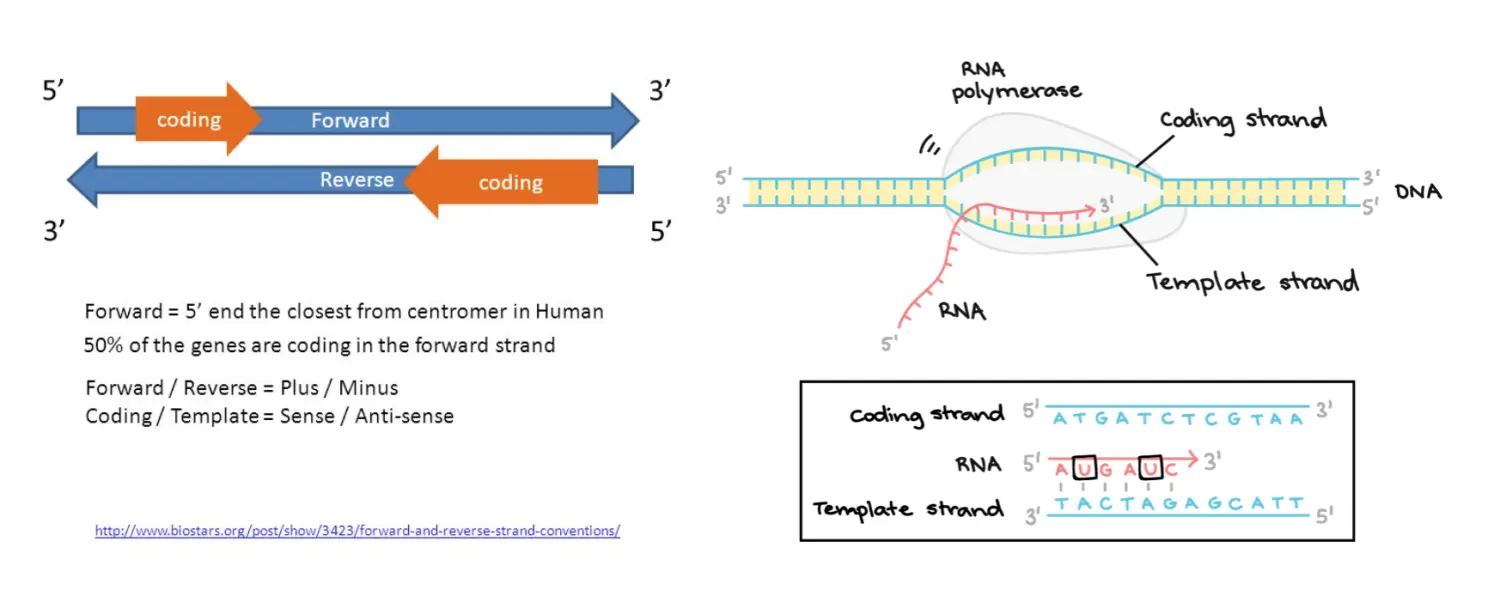
等位基因(allele):一对同源染色体相同位置上控制某一性状的不同形态的基因,可以简单想成控制同一性状的不同基因
ALT:正链上对应的改变的等位基因(可以有多个)
QUAL:
REF/ALT变异位点存在的概率(和FASTQ的质量值、SAM的MAPQ一样,都是Phred值-10 * log(1-p))FILTER:数据一般都要经过适当的过滤后才能继续使用variant callset(变异位点数据集),关于是否完成过滤会给出三种说明: 一是给出没有通过过滤的变异位点;二是
PASS表示全部通过了过滤;三是.表示这个位点没有任何过滤INFO:以
tag=value的形式给出位点注释信息;分号;分割FORMAT:针对样本的注释;冒号
:分割,并且对应后面各个sample中的信息
# 大体就是这样
#CHROM POS ID REF ALF QUAL FILTER INFO FORMAT align.bam
AF086833 60 . A T 54 . DP=43 GT:PL 0/1:51,0,48
上面是关于VCF的大体了解,下面具体看看重要的部分
具体–REF/ALT
REF表示在POS位置的参考等位基因;ALT表示在POS位置的所有变异
开始想象场景:我们用软件发现,在60bp处发现了一个变异,是A变成了T,那么应该这么表示:
#CHROM POS ID REF ALT QUAL FILTER INFO FORMAT align.bam
AF086833 60 . A T 54 . DP=43 GT:PL 0/1:51,0,48
如果同一个位置检测到有两种可能发生的变异呢?
#CHROM POS ID REF ALT QUAL FILTER INFO FORMAT align.bam
AF086833 60 . A T,C 43.2 . DP=95 GT:PL 1/2:102,124,42,108,0,48
事情还没完,刚刚两个例子都只是一个变异位点,但实际上有可能多个位点发生缺失(而这才是VCF复杂的开始),例如:58、59、60碱基(GGA)发生了缺失
#CHROM POS ID REF ALT QUAL FILTER INFO FORMAT align.bam
AF086833 55 . TGAGGA TGA 182 . INDEL;IDV=42 GT:PL 0/1:215,0,255
AF086833 60 . A C,T 39.8 . DP=126 GT:PL 1/2:99,87,47,86,0,49
这里注意:虽然我们看到缺失是从58碱基开始发生的,但是记录的是从55碱基。你会不会好奇:为什么是55而不是准确的58?为什么要表示成TGAGGA->TGA,而不是GAGGA->GA或者GGA->空 呢?这个需要引入一个新词汇”variant normalizatoin“,也就是说,所有的结果展示都是有规定的。
variant normalization: 2015年Unified representation of genetic variants文章中就论述了这个一致性标记的问题,其中写道:
A genetic variant can be represented in the Variant Call Format (VCF) in multiple different ways. Inconsistent representation of variants between variant callers and analyses will magnify discrepancies between them and complicate variant filtering and duplicate removal.
于是制定了VCF的标准化,来方便交流,规定以下几点:
用尽可能少的字母来表示变异位点;
等位基因长度不为0;
变异向左”贪婪比对“ (也就是说:一直向左比对,直到不匹配为止,然后以最左边的碱基位置表示变异的起始位置)
https://academic.oup.com/bioinformatics/article/31/13/2202/196142
因此,这里写成TGAGGA->TGA就是表示缺失位点GGA向左最远可以匹配到第55号T碱基处
具体–FORMAT
假设得到的VCF中9-11列内容如下:
#FORMAT sample1 sample2
GT:PL 0/1:51,0,48 1/1:34,32,0
表示的意思就是:在样本1中发现的变异中含有GT=0/1和PL=51,0,48这样的信息,样本2中的变异含有GT=1/1和PL=34,32,0这样的信息
看到两个名词GT和PL,那么它们是什么意思呢?
我们可以回过头去Header部分看一看,将会找到如下内容:
##FORMAT=<ID=GT, Number=1, Type=String, Description="Genotype">
##FORMAT=<ID=PL, Number=G, Type=Integer, Description="List of Phred-scaled genotype likelihoods">
还感觉看不太懂?不着急,往下接着学
具体–Genotypes
尽管我们可以根据REF和ALT知道了碱基发生了怎样的变化,但是我们想知道这个变化是只是发生在一个DNA拷贝中,还是两个拷贝都有?需要用一个指标来量化这种变异~基因型(Genotype)。GT就是用来表示样本中这个位点的基因型,其中0表示参考REF,1表示变异ALT的第一个entry,2表示ALT的第二个entry(以此类推)
对于二倍体生物,GT表示了一个样本中的等位基因:
0/0表示样本是纯合子,并且和参考的等位基因一样0/1表示样本是杂合子,有一个参考的等位基因,一个变异的等位基因1/2样本是杂合子,两个都是变异的等位基因1/1样本是纯合子,且两个都是变异的第一个等位基因2/2样本是纯合子,且两个都是变异的第二个等位基因(以此类推)
当然,如果不是二倍体,命名原理也是一样:单倍体(Haploid)只有一个GT值;多倍体有多个GT值
具体–Allele
A开头的大多和等位基因相关,如
- AC(Allele Count):该Allele数目
- AF(Allele Frequency):该Allele频率
- AN(Allel Number):Allele总数目
对于二倍体样本(Diploid sample),基因型为0/1表示杂合子:AC=1表示Allele数为1(即该位点只有1个等位基因发生变异);AF=0.5频率为0.5(该位点只有50%的等位基因发生变异);AN=2表示总Allele为2。
基因型为1/1表示纯合子,AC=2,AF=1,AN=2
正常人的二倍体基因组位点只有杂合和纯合两种情况,因此杂合AF一定是0.5,纯合AF一定是1,但实际的VCF数据是通过测序数据来的,随机性加上测序深度不够带来的系统误差,就使得结果变得不那么理想
具体–Genotype likelihoods
直白地说就是”基因型可能性“,就是用来衡量不同基因型可能发生的概率,这是利用p-value统计,因此0表示可能性最大,例如:
GT:PL 0/1:51,0,48
其中PL这一项有三个数值,分别对应三种可能的基因型(0/0,0/1,1/1)发生的概率:第一个数值51表示基因型为0/0的概率是Phred值51 ,也就是1x10^6 ;第二个数值0表示基因型为0/1的概率是0(和GT判断的一致);第三个数值48表示基因型为1/1的概率是1x10^5
具体–Allele depth and depth of coverage
软件判断是那种基因型,到底是不是发生了变异,是需要一定的统计方法的,主体就是之前比对的结果BAM文件,其中包含了reads的比对信息,这里就是根据reads比对的数量进行判断
- AD(DepthPerAlleleBySample): unfiltered allele depth 就是有多少reads出现了某个等位基因(其中也包含了没有经过variant caller过滤的reads),但是不包括没意义的reads(就是那些统计结果不过关的,没法说服软件相信这个等位基因)。在二倍体样本中表示为:逗号分隔的两个值,前者为对应的ref基因型,后者为对应alt的基因型
- DP(Coverage,即reads覆盖度,指一些reads被过滤后的覆盖度): filtered depth, at the sample level 只有通过variant caller软件过滤后的reads才能计算入内,但是DP也纳入了那些经过过滤但没有意义的reads(uninformative reads),这一点又和AD不同
具体–Genotype Quality
GQ就是用Phred值来表示GT判断的准确性,它和PL相似,但是取值不同。PL最小值0表示最准确,GQ一般取PL的第二个小的值(除非第二小的PL大于99)。在GATK分析中,GQ最大就限制在99,因为超过99其实是没有什么意义的,并且还多占字符。因此,如果GATK中发现PL值中第二个小的值比99还要大,软件就将GQ标为99。用GQ值就可以得到,第一位和第二位之间到底差了多少,因此可以快速判断分析的准不准,选择第一个靠不靠谱
Basically the GQ gives you the difference between the likelihoods of the two most likely genotypes. If it is low, there is not much confidence in the genotype.
做一个小总结–VCF帮助判断基因型
现在来看看NA12878基因(1: 899282)的统计情况
1 899282 rs28548431 C T [CLIPPED] GT:AD:DP:GQ:PL 0/1:1,3:4:26:103,0,26
这个位点的GT=0/1,可以判断基因型是C/T;GQ=26表示排名第二基因型的可能性是0.0025,结果不是很好,因为虽然判断的第一位基因型的PL 为0很可靠,但是毕竟相差不多,很难推翻第二位(如果GQ值再大一些,我们就更有信心说明判断的C/T基因型是正确的)。当然,这里GQ的原因很有可能是取样太少,只有4条reads在这个位点作为参考(DP=4),这四条中有1条带参考的碱基信息,另外3条与参考不一致,存在变异(AD=1,3)
**因此,重点的结论来啦!**尽管我们相信,这个位点确实存在变异,但是假阳性依然存在,也就意味着基因型判断结果不一定是杂合子C/T,有一定的可能是变异纯合T/T(PL(1/1)=26),但一定不可能是参考纯合C/C (PL(0/0)=103)
参考:
VCF short summary:http://www.htslib.org/doc/vcf.html
VCF Poster:http://vcftools.sourceforge.net/VCF-poster.pdf
VCF 说明书: http://samtools.github.io/hts-specs/
GATK解释VCF:https://gatkforums.broadinstitute.org/gatk/discussion/1268/what-is-a-vcf-and-how-should-i-interpret-it
Difference between QUAL and GQ annotations https://software.broadinstitute.org/gatk/documentation/article.php?id=4860
如何得到VCF?
VCF一般是利用"variant caller"或者"SNP caller"的工具对BAM比对文件操作得到的
bowtie2+samtools+bcftools
之前要生成VCF或者它的二进制BCF,需要用samtools mpileup,然后再利用bcftools去进行SNP calling。但是有一个问题就是:samtools与bcftools更新速度都很快,使用mpileup+bcftools call pipeline时会出现版本冲突导致报错的问题,于是后来直接一步到位将mpileup加入了bcftools的功能中 【关于bcftools的介绍,继续向下看】
## 下载bowtie2
cd ~/test
wget https://sourceforge.net/projects/bowtie-bio/files/bowtie2/2.3.4.3/bowtie2-2.3.4.3-linux-x86_64.zip
unzip bowtie2-2.3.4.3-linux-x86_64.zip
cd bowtie2-2.3.4.3-linux-x86_64/example/reads
## bowtie2 比对 + samtools排序
wkd=~/test/bowtie2-2.3.4.3-linux-x86_64
$wkd/bowtie2 -x $wkd/example/index/lambda_virus -1 reads_1.fq -2 reads_2.fq | samtools sort -@ 5 -o lamda.bam -
bcftools mpileup -f $wkd/example/reference/lambda_virus.fa lamda.bam |bcftools call -mv -o lamda.call.vcf
# 其中bcftools call使用时需要选择算法,有两个选项:-c(consensus-caller);-m(multiallelic-caller),其中-m比较适合多个allel与罕见变异的calling情况
# 另外-v的意思是说:只输出变异位点信息,如果位点不是SNP/InDel就不输出
GATK
# From https://www.biostars.org/p/307422/
1. trim reads
2. bwa mem align to genome
3. mark duplicates (e.g. picardtools =》MarkDuplicates)
4. use HaplotypeCaller to generate gvcf
5. CombineGVCFs
6. GenotypeGVCFs on the combined gvcf
7. filter your vcf however you want
8. You can do base recalibration iteratively now if you want with the filtered vcf.
参考:http://www.bio-info-trainee.com/3577.html
bcftools 说明书 https://samtools.github.io/bcftools/bcftools.html
拓展阅读:GATK的深度学习Convolutional Neural Networks(CNN)vs. Google DeepVariant vs. GATK VQSR https://www.biostars.org/p/301751/ [其中 Andrew认为GATK是寻找变异的金标准,但是程序有点繁琐,好用不好入门。有推荐使用samtools+bcftools的,并且它们在鉴定SNVs有优势,但InDel方面就欠缺一些]
https://gatkforums.broadinstitute.org/gatk/discussion/10996/deep-learning-in-gatk4
【!放在公众号!】小福利:2018.11最新的GATK Worksheet 链接:https://share.weiyun.com/5vefLMG 密码:yf5322
VCF基本操作
得到了VCF文件只是第一步,下面还需要一定的技巧获得想要的数据
利用bcftools
背景
bcftools与samtools同出李恒之手,是用来操作VCF文件的,之前有一个软件叫vcftools,但是就更新到2015年,bcftools是利用C语言开发,因此处理速度很快,可以接替vcftools。用过samtools的都知道,主命令下还有许多的子命令,例如:samtools index 、samtools sort、samtools flagstat等等。bcftools也是如此,主要有三大功能:
- 对VCF/BCF构建索引:
bcftools index - 对VCF/BCF进行操作(查看、排序、过滤、交集、格式转换等)
annotate、concat、convert、isec、merge、norm、plugin、query、reheader、sort、view - 找变异
call、consensus、cnv、csq、filter、gtcheck、mpileup、roh、stats
下载测试数据
# 测试文件是19号染色体:400-500kb
wget http://data.biostarhandbook.com/variant/subset_hg19.vcf.gz
gunzip subset_hg19.vcf.gz # 先解压,一会做个错误示范
构建索引 index =>需要用到bgzip格式
# 如果我们使用上面解压的subset_hg19.vcf,结果会如何呢?
$ bcftools index subset_hg19.vcf
# 报错!
index: the file is not BGZF compressed, cannot index: subset_hg19.vcf
# 因此我们需要按它的要求来压缩
$ bgzip subset_hg19.vcf
$ bcftools index subset_hg19.vcf.gz #默认是构建.csi索引
# 另外还有一种.tbi索引
$ bcftools index -t subset_hg19.vcf.gz
查看、筛选、过滤 => view
## 查看
# 同samtools一样,要想查看二进制文件(这里的.bcf),需要用view
$ bcftools view subset_hg19.bcf
# .vcf可以直接less查看
less -S subset_hg19.vcf.gz
##筛选
# 想要筛选某些样本的VCF信息(多个样本逗号分隔)
$ bcftools view subset_hg19.vcf.gz -s HG00115,HG00116 -o subset.vcf
# 如果不想要某些样本,只需要在样本名前加^
$ bcftools view subset_hg19.vcf.gz -s ^HG00115 -o subset_rm_115.vcf
##过滤
# -k参数得到已知的突变位点(ID列中不是.的那部分)
$ bcftools view subset_hg19.vcf.gz -k -o knowm.vcf
# -n参数得到位置的突变位点(可能是novel新的位点,也就是ID列中为.的部分)
$ bcftools view subset_hg19.vcf.gz -n -o unknowm.vcf
# -c参数设置最小的allele count(AC)值进行过滤(就是说某个变异位点出现了多少次)
$ bcftools view -c 5 subset_hg19.vcf.gz | grep -v "#"
标准/DIY格式转换 => view / query
## 简单的格式转换可以用view
# 将vcf转换成bcf:-O指定输出文件类型(u表示未压缩的bcf文件;b表示压缩的bcf文件;v表示未压缩的vcf文件;z表示压缩的vcf文件)
$ bcftools view subset_hg19.vcf.gz -O u -o subset_hg19.bcf
## 想定制转换后的格式,可以用query[就是从VCF/BCF中抽取出某些部分]
# 【-i:得到指定表达式的位点;-e:排除指定表达式的位点;-f:指定输出的format;-H输出表头(也就是format信息的汇总)】
##############################################################################
# 例1:想得到染色体号、变异位置、参考碱基、第一个变异碱基
$ bcftools query -f '%CHROM %POS %REF %ALT{0}\n' subset_hg19.vcf.gz
#【结果】
19 499924 T C
##############################################################################
# 例2:在例1的基础上将空格改成tab分隔,再增加样本名和基因型信息
$ bcftools query -f '%CHROM\t%POS\t%REF\t%ALT[\t%SAMPLE=%GT]\n' subset_hg19.vcf.gz
#【结果】
19 499924 T C HG00115=0|0 HG00116=0|0 HG00117=0|0 HG00118=0|0 HG00119=0|0 HG00120=0|0
##############################################################################
# 例3:做一个BED文件:chr, pos (0-based), end pos (1-based), id
$ bcftools query -f'%CHROM\t%POS0\t%END\t%ID\n' subset_hg19.vcf
#【结果】
19 499923 499924 rs117528364
##############################################################################
# 例4:将所有变异的样本以及变异情况输出
$ bcftools query -f'[%CHROM:%POS %SAMPLE %GT\n]' -i'GT="alt"' subset_hg19.vcf
#【结果】
19:499978 HG00115 0|1
19:498212 HG00118 1|1
##############################################################################
# 例5:练习-i和-H
$ bcftools query -i'GT="het"' -f'[%CHROM:%POS %SAMPLE %GT \n]' -H subset_hg19.vcf
#【结果有省略】
[1]HG00115:CHROM:[2]HG00115:POS [3]SAMPLE [4]HG00115:GT
[5]HG00116:CHROM:[6]HG00116:POS [7]SAMPLE [8]HG00116:GT
19:400666 HG00115 1|0
19:400666 HG00116 0|1
##############################################################################
# 例6:列出VCF中所有样本
$ bcftools query -l subset_hg19.vcf.gz
##############################################################################
# 例7:提取指定区域中所有的变异信息【一个region用-r;对于多个region(如BED文件)要找变异用-R】
$ bcftools query -r '19:400300-400800' -f '%CHROM\t%POS\t%REF\t%ALT\n' subset_hg19.vcf.gz | head
#【结果有省略】
19 400410 CA C
19 400666 G C
19 400742 C T
##############################################################################
# 例8:相反,如果想去掉某个区域的变异,要用-t 【-t与-r相似,都是指定一个区域,不同的是:-r跳转到那个区域,而-t是扔掉这个区域;并且-t还需要加上^】
$ bcftools query -t ^'19:400300-400800' -f '%CHROM\t%POS\t%REF\t%ALT\n' subset_hg19.vcf.gz | head
#【结果有省略】
19 400819 C G
19 400908 G T
19 400926 C T
# 思考:【如果要去掉多个区域,可以把它们放在BED文件中,然后用-T和^;有没有和例7中的-R很像?】
$ cat >exclude.bed
19 400300 400700
19 400900 401000
$ bcftools query -T ^exclude.bed -f '%CHROM\t%POS\t%REF\t%ALT\n' subset_hg19.vcf.gz | head
#【结果有省略】
19 400742 C T
19 401076 C G
19 401077 G A,T
##############################################################################
# 例9:要找到所有样本中的变异位点(也就是排除-e GT为.或者为0|0)
$ bcftools view -e 'GT="." | GT="0|0"' subset_hg19.vcf.gz | bcftools query -f '%POS[\t%GT\t]\n' | head
# 这里大括号[]的意思是:对其中的每个sample进行循环处理
#【结果有省略】
402556 0|1 0|1 1|1 1|0 0|1 1|1
402707 0|1 0|1 1|1 1|0 0|1 1|1
# TIP:如果想看是否有样本是纯合/杂合,可以用-g快速看一下【-g可以选择hom(homozygous), het(heterozygous ),miss(missing),还可以连用^表示反选】
$ bcftools view -g het subset_hg19.vcf.gz | bcftools query -f '%POS[\t%GT\t]\n' | head
# 因此,要选择所有样本都是纯合的基因型,就要排除掉het和miss
$ bcftools view -g ^het subset_hg19.vcf.gz | bcftools view -g ^miss | bcftools query -f '%POS[\t%GT\t]\n' | head
##############################################################################
# 例10:只选择InDel (顺便统计下有多少个)
$ bcftools view -v indels subset_hg19.vcf.gz | bcftools query -f '%POS\t%TYPE\n' | wc -l
# -v参数可选snps|indels|mnps|other,多选用逗号分隔;排除某个类型的变异,用-V
##############################################################################
# 例11:基于site quality(QUAL)和read depth(DP)选变异位点
$ bcftools query -i 'QUAL>50 && DP>5000' -f '%POS\t%QUAL\t%DP\n' subset_hg19.vcf.gz | head
#【结果有省略】
400410 100 7773
400666 100 8445
400742 100 15699
合并VCF/BCF => merge
用于将单个样本的VCF文件合并成一个多样本的(输入文件需要是bgzip压缩并且有.tbi索引)
# 假设有许多vcf的文件
$ cat sample.list
sp1.vcf.gz
sp2.vcf.gz
sp3.vcf.gz
...
$ bcftools merge -l sample.list > multi-sample.vcf
取交集 => isec
## 例1:假设现在有三个样本,每个样本有自己的VCF,我们现在想找它们三者之间的共同点(-p 指定输出文件的存放目录前缀;-n指定多少样本)
$ bcftools isec -p tmp -n=3 sp1.vcf.gz sp2.vcf.gz sp3.vcf.gz
## 例2:取至少两个样本中一致的变异位点
$ bcftools isec -p tmp -n+2 sp1.vcf.gz sp2.vcf.gz sp3.vcf.gz
## 例3:只选出一个样本中有,其他样本中没有的变异位点(找到距离-C最近的那个样本中特异的位点)
$ bcftools isec -p tmp -C sp1.vcf.gz sp2.vcf.gz sp3.vcf.gz
# 这样得出来的结果就是:只在sp1中存在,sp2和sp3中都没有
方便的统计=>stat
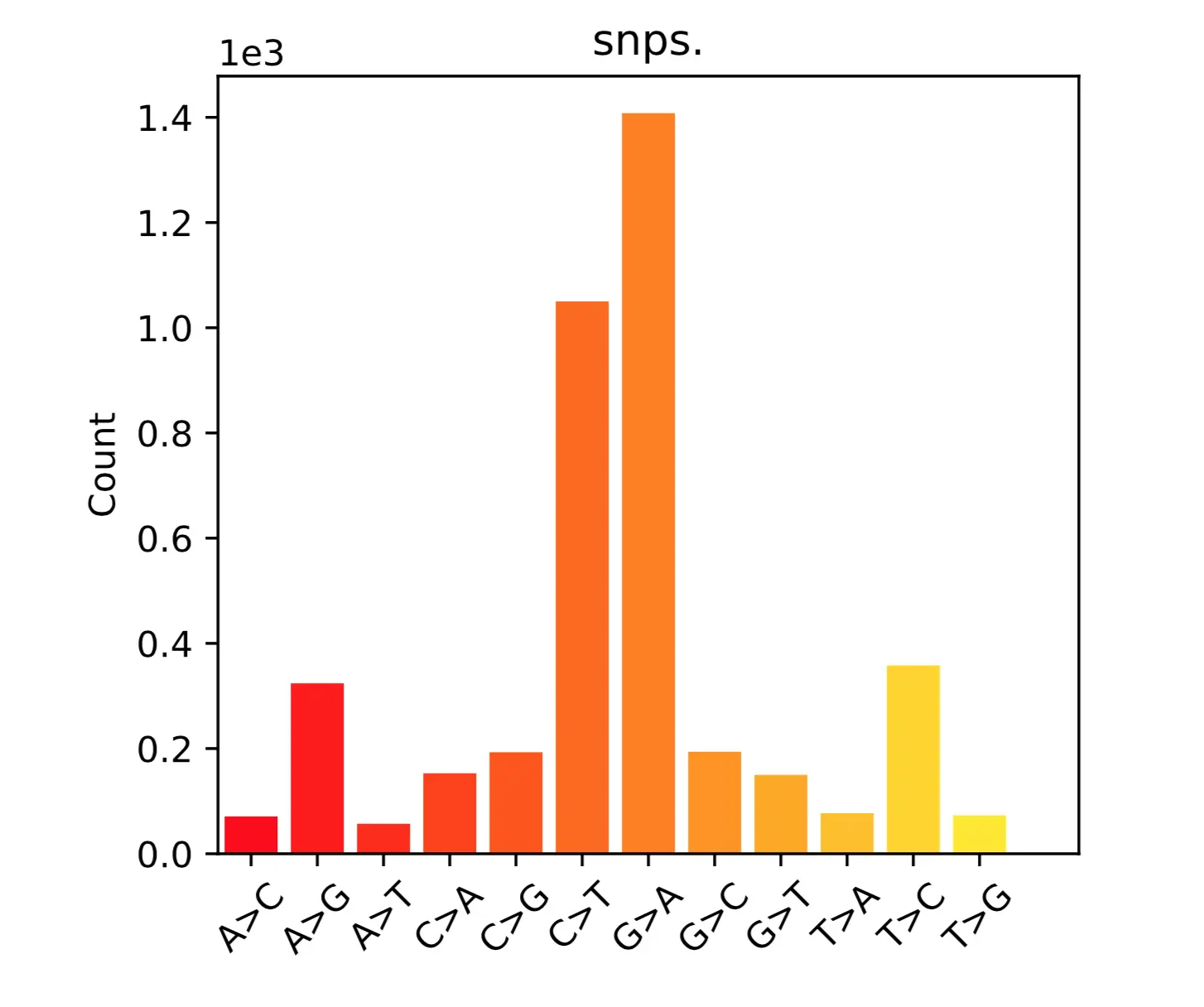
# 例如要统计SNP信息(包括)
$ bcftools view -v snps subset_hg19.vcf.gz > snps.vcf
$ bcftools stats snps.vcf > snps.stats
可视化需要安装latex、matplotlib,直接上conda,然后使用plot-vcfstats
$ plot-vcfstats snps.stats -p tmp/
# -p 指定文件夹名字,结果生成的pdf文件就存放其中。然后主要看summary.pdf
参考: bcftools的manual:https://samtools.github.io/bcftools/bcftools.html
samtools的manual:http://www.htslib.org/doc/samtools.html
bcftools examples: https://samtools.github.io/bcftools/howtos/query.html
find common variants in multiple VCF files https://justinbagley.rbind.io/2018/07/25/how-to-find-common-variants-in-multiple-vcf-files/
利用GATK
我们想要找到可靠的结果,一般会采用两种以上的软件进行分析,对于找变异也是如此。
想象几个场景:我们用不同的软件对同一个样本进行分析,然后想找到它们共同包含的变异信息;对于不同的样本,我们想知道它们有哪些相同变异,又有哪些不同变异;我们自己的样本中有没有比较特殊的变异…
GATK SelectVariants
GATK的这个功能就是用来选择VCF文件中符合指定条件的变异,它的基本用法:
# 输入参数
-V : 原始的VCF文件 [Required参数]
-select : 指定特定条件(e.g. AF<0.25; DP>1000)
-sn/sf/se : 指定筛选的样本名称/文件/正则条件
# 输出参数
-o : 筛选后的VCF文件
# 筛选设置
-ef : 去掉不符合筛选条件的变异
-env : 去掉没有变异的行
-selectType : 选择特定种类的变异(SNP, InDel ...)
-xl_{sn/sf/se}: 去掉特定的样本
输入参数中,sn(sampleName)表示单个样本名,sf(sampleFile)表示包含多个样本名的文件,se(sampleExpressions)表示用正则表示出样本名。
需要注意的是:SelectVariants功能只是标出了符合条件的变异,并不会默认去除不符合条件的变异。
如果只想保存符合条件的变异,使用ef(excludeFilteredVariants) ;
如果从多个样本中选取变异信息,但其实它们都存在共同的没有变异的位点,就可以用env(excludeNonVariants)去除;
利用ef与env,可以去除不是这个人种的变异位点
例如:我们想看看自己的样本中有多少变异存在于1000Genome中
java -jar GATK.jar \
-R reference.fasta \
-T SelectVariants \
-V 1000G_ALL_Genotypes.vcf.gz \
-sf samples.list \ # 将文件名都存在这个list中
-ef \ #--excludeFiltered 保留PASS与.的位点
-env \ #去除样本中没有在1000Genome中出现的位点
-o 1000G_.vcf \
# 最后生成的文件中只包含过滤没有fail并且至少一个样本中有变异的位点
更详细的参数说明参考官方:https://software.broadinstitute.org/gatk/documentation/tooldocs/3.8-0/org_broadinstitute_gatk_tools_walkers_variantutils_SelectVariants.php
GATK CombineVariants
上面是说提取符合特定条件的子集,许多时候还需要合并多个VCF文件。当合并多个文件时,如果包含同一个位点,但位点信息不一样,就需要考虑设置合并参数。基本用法:
# 输入参数
-V : 多个vcf文件(多个文件可以添加tag描述,用于区分)
# 可选参数
-genotypeMergeOptions :#关于合并同一个样本的不同基因型
-filteredAreUncalled : #忽略filtered变异位点
-filteredRecordsMergeType : #如何对待filter status不一样的位点
-priority #对于多个vcf文件,设置它们的基因型优先级(需要配合之前添加-V时的tag描述,逗号分隔,来表示不同的文件)
# 输出参数
-o :
genotypeMergeOptions的设置:如果目的是Merge two separate callsets,就可以用UNIQUIFY;如果目的是Get the union of calls made on the same samples,就可以用PRIORITIZE
filteredRecordsMergeType的设置:KEEP_IF_ANY_UNFILTERED是至少有一个filter status不一致就不合并这个位点;KEEP_IF_ALL_UNFILTERED是当所有的status不一致才不合并这个位点;KEEP_UNCONDITIONAL不管filter status,只要是位点信息,就合并
例如,我们有两个vcf文件需要合并
java -jar GATK.jar \
-R reference.fa \
-T CombineVariants \
-V:hua file1.vcf \ # 如果文件比较多,可以写到list中,然后只用-V file.list
-V:dou file2.vcf \
-filteredAreUncalled \
-o output.vcf
# 结果文件中,包含了两个输入文件中的变异,每个变异位点都有一个"set=="栏,标识位点来源,可以来自file1可以来自file2,或者来自二者
官方说明:https://software.broadinstitute.org/gatk/documentation/tooldocs/3.8-0/org_broadinstitute_gatk_tools_walkers_variantutils_CombineVariants.php
Combine联合Select
例如,我们想找到某个病人特有变异
java -jar GATK.jar \
-R reference.fa \
-T CombineVariants \
-V:1000G 1000G_ALL_Sites.vcf \ #ESP6500 SNP文件
-V:ESP ESP6500.snps.vcf \ #1000G数据库文件
-V:D2D D2D.vcf \ #假设使我们得到的vcf文件
-o patient.D2D.combined.vcf # 合并的文件
java -jar GATK.jar \
-R reference.fa \
-T SelectVariants \
-V patient.D2D.combined.vcf \ # 加载合并好的vcf文件
-select "set==D2D" \ # 选择标记的D2D位点
-o patient.D2D.private.vcf #仅在该病人中有,在ESP6500和1000G中没有的变异位点
找到变异,然后呢?
变异位点的注释
我们得到变异位点,但仅仅是知道了它们在基因组上的位置信息和相关的碱基信息。那么还存在许许多多的疑问没有解决:
这个位点是在基因上吗?是内含子还是外显子区域?这个突变对基因功能产生了什么影响?对于转录翻译有没有影响?除了研究的样本,还有没有其他样本也出现了这个变异?有的话是什么人种,又是什么病例?
这些问题都要靠变异注释来解决
一般来说,变异注释分为:突变频率注释、变异的蛋白功能危害注释、剪切位点突变危害注释、突变相关的疾病注释
突变频率注释
做这个内容的数据库有许多,其中比较重要的有dbSNP、1000人基因组项目(1000 Genome)、ExAC、gnomeAD
dbSNP(The single-nucleotide polymorphism database):http://www.ncbi.nlm.nih.gov/SNP/ NCBI与人类基因组研究所合作建立,包含了SNP、短重复序列、微卫星标记等来源、检测方法、基因型信息、上下游序列、人群分布频率等
1000G (千人基因组项目) 研究时限:2008-2015年;汇集30个人种、3904个样本WGS和WES测序结果。目前已被ANNOVAR收纳为变异位点在正常人群中进行突变频率注释的数据库,实际分析中也应该将1000G的不同人群作为control组进行疾病关联分析
它的构建总共分为4个阶段: 一、Pilot phase 【A map of human genome variation from population-scale sequencing Nature 467, 1061–1073 (28 October 2010)】

二、Phase one 【An integrated map of genetic variation from 1,092 human genomes Nature 491, 56–65 (01 November 2012)】
三、Phase two
四、Phase three 【A global reference for human genetic variation Nature 526, 68–74 (01 October 2015); An integrated map of structural variation in 2,504 human genomes Nature 526, 75–81 (01 October 2015)】
参考:http://www.internationalgenome.org/about
含有两个下载镜像:ftp://ftp.1000genomes.ebi.ac.uk/vol1/ftp/
ftp://ftp-trace.ncbi.nih.gov/1000genomes/ftp/
其中Phase3所有的样本信息下载:ftp://ftp.1000genomes.ebi.ac.uk/vol1/ftp/technical/working/20130606_sample_info/
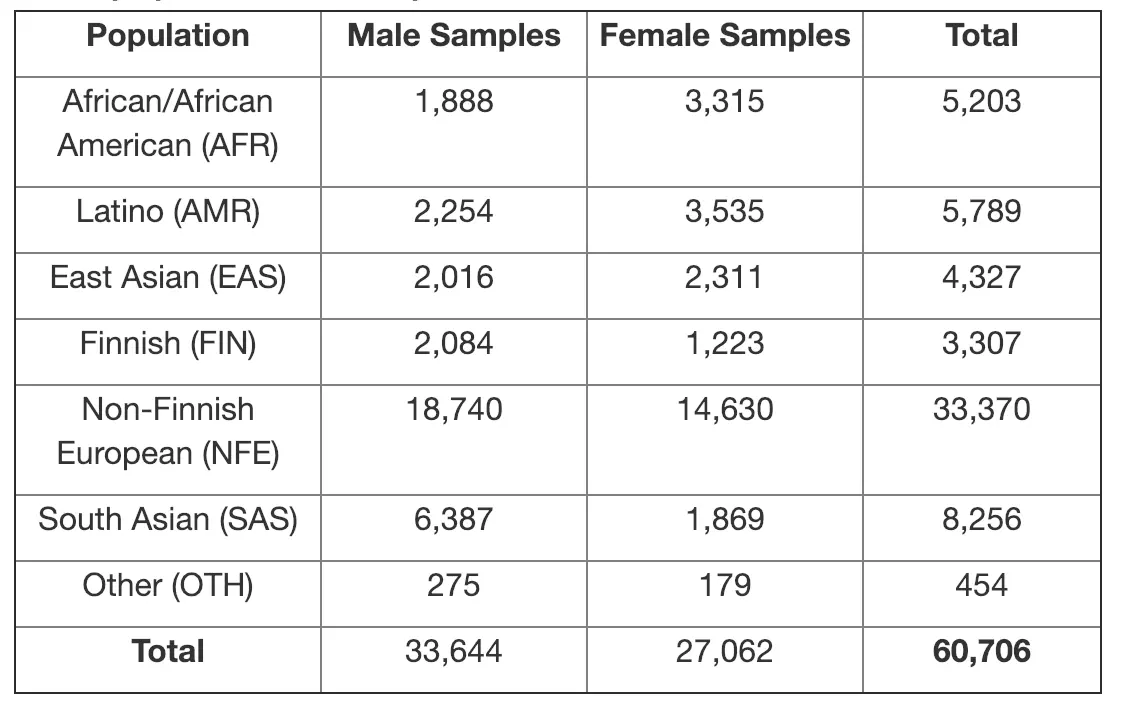
ExAC(Exome Aggregation Consortium):整合了60706个人的WES测序数据及相关遗传信息,包含超过1000万种基因变异信息 http://exac.broadinstitute.org/ 。包括了AFR(African)、AMR(Admixed American)、EAS(East Asian)、FIN(Finnish)、NFE(NON-finnish European)、SAS(South Asia)等种群的突变频率(AF)信息http://exac.broadinstitute.org/faq
gnomeAD:(Genome Aggregation Database)博得研究所支持建立,包含了千人基因组、ESP数据库以及绝大部分的ExAC数据库。目前有125,748个外显子数据和15,708个基因组数据 http://gnomad.broadinstitute.org/,这些数据来自大型人群测序和疾病研究项目
变异的蛋白功能危害注释
- PROVEAN:(Protein Variation Effect Analyzer)http://provean.jcvi.org/index.php 用来预测SNP或者InDel是否影响蛋白质的生物功能,不仅可以对CDS区域的非同义突变进行预测,还可以对CDS区域的非移码InDel对蛋白功能的影响进行预测,并将结果大致分为:危害、可以容忍、无害
- SIFT:(Sorting Intolerant From Tolerant)https://sift.bii.a-star.edu.sg/ 根据氨基酸在蛋白序列中的保守程度来预测氨基酸的变化对蛋白功能造成的影响。其中保守程度是比对进化关系较近的蛋白序列得到,分值(SIFT-score)表示突变对蛋白序列的影响,分值越小越严重 ,一般认为:SIFT值小于0.05为有害(D:Deleterious),大于0.05表示容忍(T:Tolerance)
- Polyphen2_HAVR: (Polymorphism Phenotyping v2) http://genetics.bwh.harvard.edu/pph2/dokuwiki/downloads 根据HumanVar数据库预测突变对蛋白的影响,来诊断孟德尔遗传病。分值表示SNP导致蛋白结构或功能改变的可能性,越大越严重
- Polyphen2_HDIV: 根据HumanDiv数据库预测**,分值越大越严重**
- LRT:也是基于序列保守性进行预测(像SIFT和Polyphen)http://www.genetics.wustl.edu/jflab/lrt_query.html 。对每一个测试的密码子,LRT将来自31个物种的氨基酸进行比对来预测突变的危害。结果的**有害突变(D:Deleterious)**表示:突变来自高度保守的密码子;突变氨基酸在其他比对的真核哺乳动物中不存在。**中性突变(N: Neutral)**表示:突变发生在非高度保守的密码子;突变的氨基酸至少在一个进行比对的真核哺乳动物中发现
剪切位点突变危害注释
如果突变发生在剪切位点附近,我们可以判断它对剪切的危害。可以用的软件有:DbscSNV、Spidex、MaxEntScan
DbscSNV:属于VEP(Variant Effect Predictor)插件,http://asia.ensembl.org/info/docs/tools/vep/script/vep_plugins.html#plugins_existing
由AdaBoost与Random Forest开发,它根据突变前后分值的变化来预测剪切位点的突变危害性
Spidex:http://www.openbioinformatics.org/annovar/spidex_download_form.php 基于深度学习,因此预测的剪切变异可能距离常规的剪切位点比较远(这一点和DbscSNV不同)
MaxEntScan:对5’剪切位点附近的6bp内含子与3bp的编码区(http://genes.mit.edu/burgelab/maxent/Xmaxentscan_scoreseq.html)以及3‘ 剪切位点附近20bp的内含子与3bp的编码区内突变进行预测(http://genes.mit.edu/burgelab/maxent/Xmaxentscan_scoreseq_acc.html),按照突变前后分值变化来得到结论(认为有危害:突变后比突变前分值降低15%以上)
突变相关的疾病注释
OMIM:(Online Mendelian Inheritance in Man)https://www.omim.org/在线人类孟德尔遗传信息数据库,包含了遗传性的基因疾病信息与表型信息,目前收录了16000多个基因词条和5400多表型词条
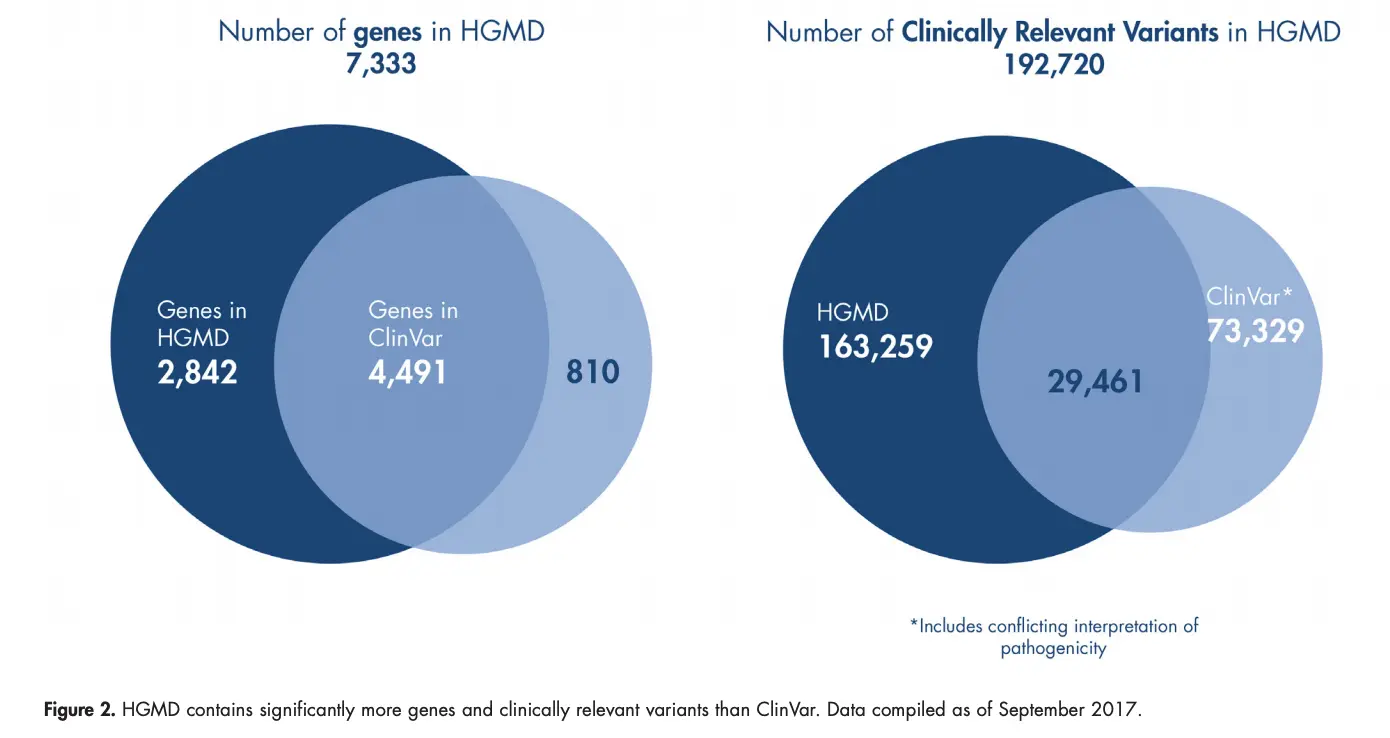
HGMD:(The Human Gene Mutation Database)1996年创立的人类基因突变数据库,目前包括240,269个变异,覆盖9976个基因。收集的突变包含了SNP、InDel、CNV、SV、基因重组等,可以说是遗传病变异检测金标准数据库。有两个版本,一个是免费的学术public版,但更新慢(http://www.hgmd.cf.ac.uk/ac/index.php);另一个是收费可试用的Professional版(https://www.qiagenbioinformatics.com/products/human-gene-mutation-database/),包含的变异数量也更多
ClinVar:2013年创立,是一个已报道突变与疾病表型关联数据库,https://www.ncbi.nlm.nih.gov/clinvar/。数据主要来源是OMIM、dbSNP、locus specific database等开源数据库,对变异位点的审核比较缺乏,因此会包含报道中冲突的致病位点
练一个工具–snpEff
conda安装
$ conda install -y snpeff
看看snpeff目前有什么数据库
# 目前有42791个数据库
$ snpEff databases > listing.txt
找到Ebola相关数据库
$ cat listing.txt | grep Homo_sapiens
#GRCh37.75 Homo_sapiens http://downloads.sourceforge.net/project/snpeff/databases/v4_3/snpEff_v4_3_GRCh37.75.zip
下载数据库
$ snpEff download GRCh37.75
# 或者
$ wget -c http://downloads.sourceforge.net/project/snpeff/databases/v4_3/snpEff_v4_3_GRCh37.75.zip
进行注释
$ snpEff GRCh37.75 subset_hg19.vcf > subset_hg19.anno.vcf
结果
主要还是看官方manual,得到的新注释的vcf中最明显的变化就是INFO列增加了一个字段ANN,默认ANN中又会给出几种信息
Allele:列出突变碱基
Annotation:列出
Sequence Ontology中的条目,表示变异的后果或者影响(effect or consequence),例如intron_variant;如果有多个,用&连接intron_variant&nc_transcript_variantPutative impact:变异位点的危害程度大小,四个取值:HIGH、MODERATE、LOW、MODIFIER

Gene name:HGNC官方基因名
Gene ID
Feature type:feature信息(如transcript, motif, miRNA等等),如果是组织特异性信息,可以添加细胞类型或者组织信息等(用冒号隔开),如
H3K4me3:HeLa-S3Feature ID :根据type来决定ID,比如type是transcript,那么就是Transcript ID,另外还有Motif ID、miRNA、ChipSeq peak、Histone mark等
Transcript biotype:Ensembl数据库的转录本类型(
Coding / Noncoding)Rank: 变异位点出现在基因区域时,会给出位点在
exon/intron的第几位。例如,变异位点出现在某基因的第2个exon上,而这个基因共有10个exon,因此就显示2/10HGVS.c:根据HGVS(http://www.hgvs.org/)标准命名的基因水平变异
HGVS.p:根据HGVS标准命名的蛋白水平变异(前提是变异位点在编码区)。如果Transcript ID在feature ID中表示出来了,这里就可以省略
cDNA_position(可选cDNA_len):变异位点在cDNA的位置(或cDNA的总长度)
CDS_position (可选CDS_len):变异位点在CDS的位置(或CDS长度)
Protein_positoin (可选Protein_len):位点在AA的位置(或AA总长度)
Distance:变异位点与最接近的feature的距离,例如位点在exon,会给出与最近的内含子的距离;位于基因间区会给出与最近基因的距离

Errors,Warnings: 注释可靠性评估【见官网】
参考:snpEff manual http://snpeff.sourceforge.net/VCFannotationformat_v1.0.pdf
http://snpeff.sourceforge.net/SnpEff_manual.html
附录1:VCF主体部分介绍
CHROM [chromosome]: 染色体名称。
POS [position]: 参考基因组突变碱基位置,如果是INDEL(插入缺失),位置是INDEL的第一个碱基位置。
ID [identifier]: 突变的名称。若没有,则用‘.’表示其为一个新变种。
REF [reference base(s)]: 参考染色体的碱基,必须是ATCGN中的一个,N表示不确定碱基。
ALT [alternate base(s)]: 与参考序列比较,发生突变的碱基;多个的话以“,”连接, 可选符号为ATCGN*,大小写敏感。
QUAL [quality]: Phred标准下的质量值,表示在该位点存在突变的可能性;该值越高,则突变的可能性越大;计算方法:Phred值 = -10 * log (1-p) p为突变存在的概率。
FILTER [filter status]: GATK使用其它的方法进行过滤后得到的过滤结果,如果通过则该值为“PASS”;若此突变不可靠,则该项不为”PASS”或”.”。
INFO [additional information]: 表示变异的详细信息
DP [read depth]: 样本在这个位置的一些reads被过滤掉后的覆盖度
DP4 : 高质量测序碱基,位于REF或者ALT前后
MQ [mapping quality]: 表示覆盖序列质量的均方值RMS
FQ : Phred值关于所有样本相似的可能性
AF [allele frequency]: 表示Allele(等位基因)的频率,AF1为第一个ALT等位基因发生频率的可能性评估
AC [allele count]: 表示Allele(等位基因)的数目,AC1为对第一个ALT等位基因计数的最大可能性评估
AN [allele number]: 表示Allele(等位基因)的总数目
IS : 插入缺失或部分插入缺失的reads允许的最大数量
AC [allele count]: 表示该Allele(等位基因)的数目
G3 : ML 评估基因型出现的频率
HWE : chi^2基于HWE的测试p值和G3
CLR : 在受到或者不受限制的情况下基因型出现可能性的对数值
UGT : 最可能不受限制的三种基因型结构
CGT : 最可能受限制三种基因型结构
PV4 : 四种P值的误差,分别是(strand、baseQ、mapQ、tail distance bias)
INDEL : 表示该位置的变异是插入缺失
PC2 : 非参考等位基因的Phred(变异的可能性)值在两个分组中大小不同
PCHI2 : 后加权chi^2,根据p值来测试两组样本之间的联系
QCHI2 : Phred标准下的PCHI2.
PR : 置换产生的一个较小的PCHI2
QBD [quality by depth]: 表示测序深度对质量的影响
RPB [read position bias]: 表示序列的误差位置
MDV : 样本中高质量非参考序列的最大数目
VDB [variant distance bias]: 表示RNA序列中过滤人工拼接序列的变异误差范围
GT [genotype]: 表示样品的基因型。两个数字中间用|分 开,这两个数字表示双倍体的sample的基因型。
0 表示样品中有ref的allele
1 表示样品中variant的allele
2表示有第二个variant的allele
0/0 表示sample中该位点为纯合的,和ref一致
0/1 表示sample中该位点为杂合的,有ref和variant两个基因型
1/1 表示sample中该位点为纯合的,和variant一致
GQ [genotype quality]: 表示基因型的质量值。Phred格式的质量值,表示在该位点该基因型存在的可能性;该值越高,则Genotype的可能性越 大;计算方法:Phred值 = -10 * log (1-p) p为基因型存在的概率。
GL : 三种基因型(RR RA AA)出现的可能性,R表示参考碱基,A表示变异碱基
DV : 高质量的非参考碱基
SP : Phred的p值误差线
PL [provieds the likelihoods of the given genotypes]: 指定的三种基因型的质量值。三种指定的基因型为(0/0,0/1,1/1),这三种基因型的概率总和为1。该值越大,表明为该种基因型的可能性越小。 Phred值 = -10 * log (p) p为基因型存在的概率。
FORMAT : 用于描述样本的(可选)可扩展的字段列表
SAMPLEs : 对于文件中描述的每一个(可选)样本,给出了在格式中列出的字段的值
附录2:vcf练习题
http://www.bio-info-trainee.com/3577.html
找到基因型不是
1/1的条目,个数$ bcftools view -e 'GT="1|1"' subset_hg19.vcf.gz | wc -l突变记录的vcf文件区分成 INDEL和SNP条目
# 得到INDEL $ bcftools view -v indels subset_hg19.vcf.gz > indel.vcf # 得到SNP $ bcftools view -v snps subset_hg19.vcf.gz > snp.vcfINDEL条目再区分成insertion和deletion统计
$ conda install bedops -y $ vcf2bed --insertions < subset_hg19.vcf | wc -l # insertion $ vcf2bed --deletions < subset_hg19.vcf | wc -l # deletion # 用命令行的话:一开始想的是这样[但事实证明想简单了,下面进行说明] awk '! /\#/' subset_hg19.vcf | awk '{if(length($4) > length($5) ) print}' | wc -l # 得到insertion awk '! /\#/' subset_hg19.vcf | awk '{if(length($4) < length($5) ) print}' | wc -l #得到deletion这里发现一个问题:同样得到deletion的结果,但是软件vcf2bed计算的结果比awk计算的要少。我特意找了一下它们的结果做了对比,发现问题出在原始vcf文件中:原始文件中的INDEL有这样一种情况

在406852位点原来的TC对应两个变异,一个是Insertion,一个是Deletion,那么软件vcf2bed在处理的时候,既算了insertion一次,又算了deletion一次;
但是用命令行awk自己做,就不能算进来这样的数据,说明一开始没有考虑到这种情况。后来找到另一个命令行处理https://bioinformatics.stackexchange.com/questions/769/how-can-i-extract-only-insertions-from-a-vcf-file
zcat subset_hg19.vcf.gz | perl -ane '$x=0;for $y (split(",",$F[4])){$x=1 if length($y)>length($F[3])}print if /^#/||$x' | grep -v "#" | wc -l #得到insertion关于一行对应多个突变的情况,这是正常的。因为VCF是一个位点描述文件,原则上可以描述多个突变(包括野生型非突变allele),因此可以有多个插入、缺失、单核苷酸SNV的存在【参考:https://anjingwd.github.io/AnJingwd.github.io/2018/01/20/ANNOVAR%E8%BF%9B%E8%A1%8C%E7%AA%81%E5%8F%98%E6%B3%A8%E9%87%8A/】
组合筛选变异位点质量值大于30并且深度大于20的条目
bcftools query -i 'QUAL>30 && DP>20' -f '%POS\t%QUAL\t%DP\n' subset_hg19.vcf.gz | head看一下总体质量值分布【来自直播基因组-27】
# 根据第5列QUAL perl -alne '{next if/^#/;$tmp=int($F[5]/10);$h{$tmp}++}END{print "$_\t$h{$_}" foreach sort {$a <=> $b} keys %h}' subset_hg19.vcf >vcf.quality.stat # 因为是部分测试数据,所以全部的QUAL都为100,但是真实情况有许多10、20的,一般低于20的需要舍弃统计测序深度的分布
perl -alne '{next if/^#/; /DP=(\d+);/;$tmp=$1;next unless $tmp;$tmp=int($tmp/10);$h{$tmp}++;}END{print "$_\t$h{$_}" foreach sort {$a <=> $b} keys %h}' subset_hg19.vcf >vcf.depth.stat看下杂合与纯合变异位点分布
$ grep INDEL subset_hg19.vcf |perl -alne '{@tmp=split/:/,$F[9];print $tmp[0]}' |sort |uniq -c
