scRNA-单细胞转录组学习笔记-5
刘小泽写于19.6.17 笔记目的:根据生信技能树的单细胞转录组课程探索smart-seq2技术相关的分析技术 课程链接在:http://jm.grazy.cn/index/mulitcourse/detail.html?cid=53 第二单元第三讲:得到表达矩阵后应该再怎么操作
过滤后的操作
上次得到的dat表达矩阵过滤掉低表达基因后,剩下12198个基因
看看其中的spike-in情况
> grep('ERCC',rownames(dat))
[1] 12139 12140 12141 12142 12143 12144 12145 12146 12147 12148 12149 12150
[13] 12151 12152 12153 12154 12155 12156 12157 12158 12159 12160 12161 12162
[25] 12163 12164 12165 12166 12167 12168 12169 12170 12171 12172 12173 12174
[37] 12175 12176 12177 12178 12179 12180 12181 12182 12183 12184 12185 12186
[49] 12187 12188 12189 12190 12191 12192 12193 12194 12195 12196 12197 12198
关于ERCC可以看这篇文章:Power Analysis of Single Cell RNA-Sequencing Experiments http://biorxiv.org/content/early/2016/09/08/073692
另外还有一篇文献讲ERCC的评价:Evaluation of the External RNA Controls Consortium (ERCC) reference material using a modified Latin square design https://bmcbiotechnol.biomedcentral.com/articles/10.1186/s12896-016-0281-x
以及这一篇:究竟什么是spike-in,怎么用它?The Overlooked Fact: Fundamental Need for Spike-In Control for Virtually All Genome-Wide Analyses https://mcb.asm.org/content/36/5/662
小故事
2003年一个负责制定科技标准的组织National Institute of Standards and Technology (NIST) 开了一个会,想要设立通用的RNA参考物,在基因表达定量时可以使用(就是怎么去判断基因高表达还是低表达,需要一个标准线)。然后它资助并参与建立了External RNA Controls Consortium (ERCC) 这个联盟,目的就是干这件事。ERCC成立后,做了一个事就是:测了176个可以转录的DNA序列作为一个对照文库(在这篇文献有介绍:https://bmcgenomics.biomedcentral.com/articles/10.1186/1471-2164-6-150),编号**ERCC-00001到ERCC-00176。**利用芯片和荧光定量进行评价和筛选,看看实验和测序平台得到的定量结果是否一致,最终筛选出96个序列。每一条序列可以保证在一次实验的所有样本中的表达量都是一致的。
好,那么它究竟是什么?
https://www.thermofisher.com/order/catalog/product/4456740 首先要知道spike-in是已知浓度的外源RNA分子。在单细胞裂解液中加入spike-in后,再进行反转录。最广泛使用的spike-in是由External RNA Control Consortium (ERCC)提供的。目前使用的赛默飞公司提供的ERCC是包括92个不同长度和GC含量的细菌RNA序列,因此它和哺乳动物转录组不同,主要体现在转录本长度、核苷酸成分、polyA长度、没有内含子、没有二级结构。polyA尾大约15nt(一般保守的内源mRNA的polyA尾有250nt)。用它是为了更好地估计和消除单细胞测序文库的系统误差(除此以外,还有一种UMI在10X中常用)。ERCC应该在样本解离后、建库前完成添加。
可能你会想,ERCC是内参基因吗? 其实并不是,它相对于内参基因会更稳定,看:https://cofactorgenomics.com/6-changes-thatll-make-big-difference-rna-seq-part-3/ Spike-in controls are inherently advantageous to endogenous housekeeping genes for normalization, as potential housekeeping genes such as ACTB, GAPDH, HPRT1, and B2M, etc. vary considerably under different experimental conditions
能干什么?
评价准确性Accuracy:定量结果和已知的spike-in相关性如何
评价敏感性Sensitivity:最少需要多少数量的RNA分析才能检测到spike-in的存在
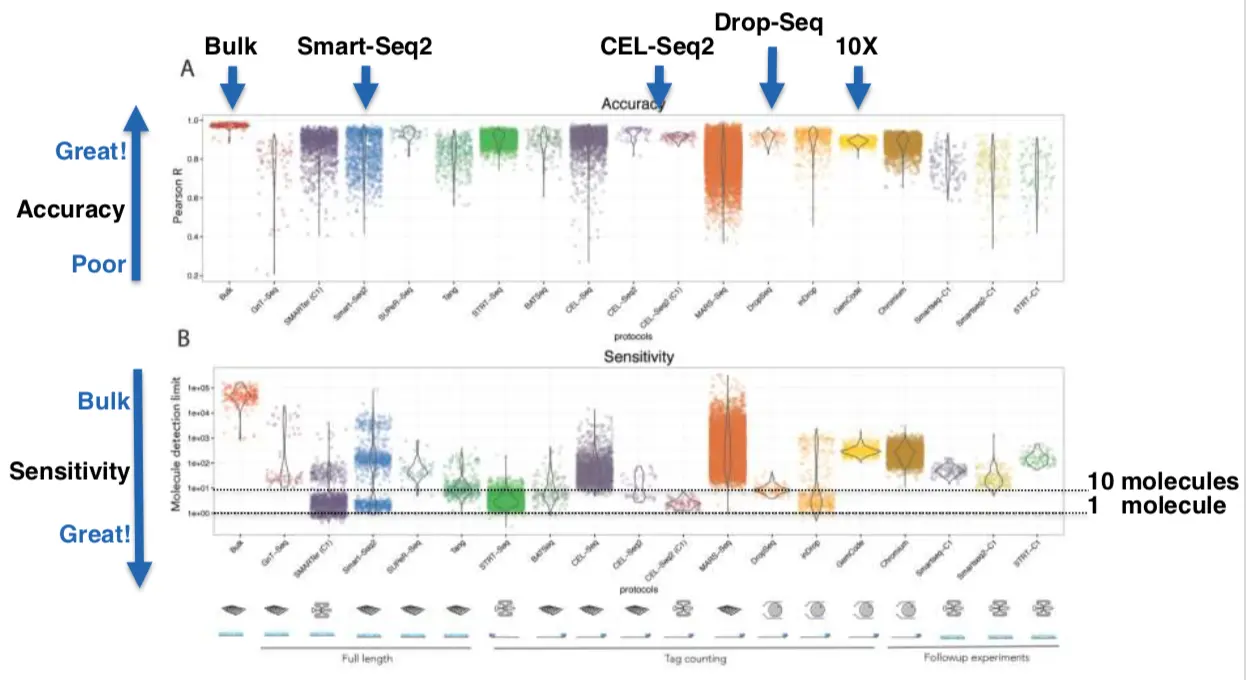
在这篇文章中(https://f1000research.com/posters/6-434#),提到了:加入的ERCC保持一个浓度,在这个浓度下,如果有超过50%的ERCC在所有样本中都能检测到,就说明这个基因可以被检测到。
高ERCC含量与低质量数据相关,通常是排除的标准。(ftp://ftp.sanger.ac.uk/pub/resources/theses/aak/chapter3.pdf)这本书中第97页写到:Each cell was spiked with exactly the same amount of ERCCs and thus the ratio of reads mapping to ERCCs to reads mapping to all mouse genes depends only on the amount of transcripts in the cell and the higher it is the lower mRNA content of the cell.
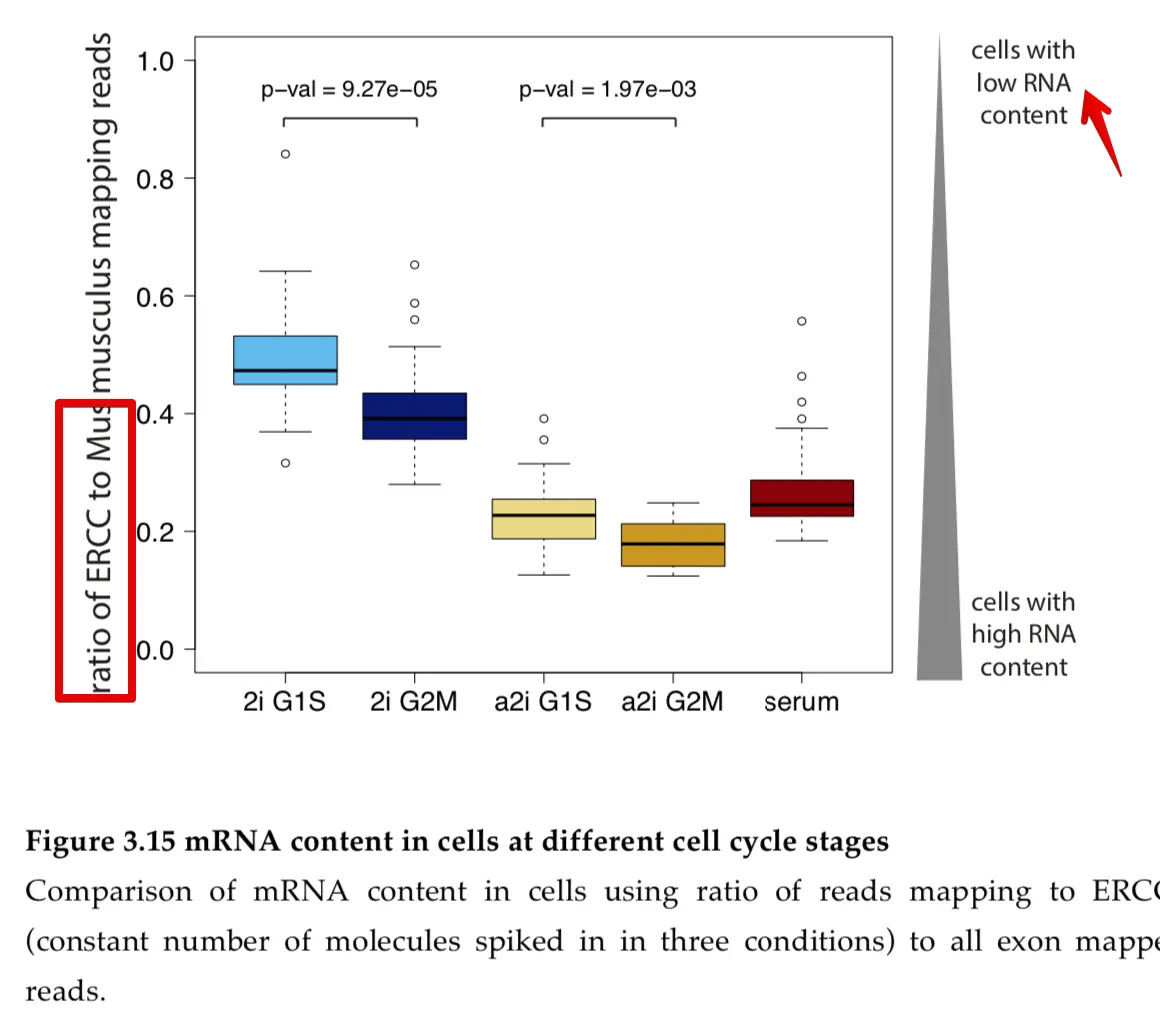
另外https://scrnaseq-course.cog.sanger.ac.uk/website/cleaning-the-expression-matrix.html也有提及:

如果ERCC的reads数很高,则表示起始内源性RNA总量低(可能发生了细胞凋亡或者其他胁迫因素导致的RNA降解;另外还可能是细胞体积小,一般来讲小细胞比大细胞有更高比例的ERCC)。
存在的问题:
其实是否要加spike-in目前还是存在争议的:Spike-ins的使用浓度通常很高,因此会占据很大比例的测序reads;ERCC的捕获效率要低于内源mRNA( Svensson et al., 2017);ERCC会显示高的技术误差,某些情况下会比内源mRNA的表达量更高;另外spike-in的定量会受生物学因素的影响,这会影响它作为对照的可信度
去除细胞文库大小差异
每个细胞测得数据大小不同,这样是没办法看高表达还是低表达的,必须先保证基数一样才能比较,cpm(counts per million)这个算法就是做这个事情的。
cpm是归一化的一种方法,代表每百万碱基中每个转录本的count值
注意:这个算法只是校正文库差异,而没有校正基因长度差异。要注意我们分析的目的就是:比较一个基因在不同细胞的表达量差异,而不是考虑一个样本中不同两个基因的差异,因为"没有两片相同的树叶”这个差异是正常的。但是同一个基因由于某种条件发生了改变,背后的生物学意义是更值得探索的。
用起来很简单,有现成的函数cpm() ,然后我们再用log将数据降个维度,但保持原有数据形状不变:log2(edgeR::cpm(dat)+1)
意思就是:cpm需要除以测序总reads数,而这个值作为分母会导致结果千差万别,有的特别大有的很小。为了后面可视化不受极值的影响,用log转换一下可以将数值变小,并且原来大的数值最后还是大,并不改变这个现实
那么具体这个函数做了什么事,才是真正需要了解的:
# 先看看前4行4列的数据
> dat[1:4,1:4]
SS2_15_0048_A3 SS2_15_0048_A6 SS2_15_0048_A5 SS2_15_0048_A4
0610007P14Rik 0 0 18 11
0610009B22Rik 0 0 0 0
0610009L18Rik 0 0 0 0
0610009O20Rik 0 0 1 1
# 比如先计算一下第三个样本的总测序量
> sum(dat[,3])
[1] 206831 #结果是0.2M
# 那么对于第三个样本SS2_15_0048_A5的第一个基因0610007P14Rik(结果是18)
# 计算它的cpm值:count值*1000000/总测序reads
> 18*1000000/206831
[1] 87.02757
# 和标准公式比较看看,结果完全相同
> edgeR::cpm(dat[,3])[1]
[1] 87.02757
# 因此最后就是
dat=log2(edgeR::cpm(dat)+1)
归一化后聚类
第一步:理解dist函数
首先理解,它是计算距离用的,正如函数名称所描述的一样:distance
# 先构建一个测试矩阵
x=1:5
y=2*x
z=52:56
tmp=data.frame(x,y,z)
> tmp
x y z
1 1 2 52
2 2 4 53
3 3 6 54
4 4 8 55
5 5 10 56
# 可以看到,x和y是有一定相关性的,而z和它们很难扯上关系
# 然后尝试计算x、y、z之间的距离,来验证我们的猜想
> dist(tmp)
1 2 3 4
2 2.449490
3 4.898979 2.449490
4 7.348469 4.898979 2.449490
5 9.797959 7.348469 4.898979 2.449490
# 好像得到的不是我们想要的。我们想要的是x、y、z距离结果,而计算给出的是以"行"为单位的结果
# 因此,猜测dist应该是以行为输入。因此修改一下tmp,让x、y、z为行,其实也就是转置一下,转置函数用t()
> dist(t(tmp))
x y
y 7.416198
z 114.039467 107.377838
同样的,我们这里的dat数据,是要计算细胞间的距离,也就是列与列之间的距离,使用dist(t(dat)) 计算。数据中有768个细胞,也就是要计算768个细胞核768个细胞之间的距离,计算量还是很大的。
关于dist计算距离的方法:主要有6种:”欧式euclidean”, “切比雪夫距离maximum”, “绝对值距离manhattan”, “Lance距离canberra”, “定型变量距离binary” or “明可夫斯基距离minkowski(使用时要指定p值)”。
默认使用第一种欧氏距离,它计算的是:几何空间中两点之间的距离。思想类似于勾股定理求第三条斜边的长度=》平方和再开方。
第二步:理解hclust函数
它是进行层次聚类(系谱聚类)的方法
关于hclust聚类的方法:”离差平方和法ward”, “最短距离法single”, “最长距离法complete”,”类平均法average”, “相似法mcquitty”, “中间距离法median” or “重心法centroid”。默认使用complete算法
hc=hclust(dist(t(dat)))
# 如果要进行可视化
plot(hc,labels = FALSE) #labels这个选项的意思是不显示各个样本名称,因为样本太多,会让图看起来很乱
另外hclust函数还有一个亲戚:cutree,顾名思义,就是对聚类树进行修剪。我们知道聚类结果是分群的,cutree就是指定输出哪些群(结果是从大群到小群排列)
# 例如要看看分的4大群
clus = cutree(hc, 4)
group_list= as.factor(clus) #得到的这个因子型变量group_list中样本顺序和输入的顺序一致,并且属于第几类都有记录
> table(group_list)
group_list
1 2 3 4
312 300 121 35
提取批次信息
在上一步操作结果中,可以看到,样本名都是有规律的,例如:
> head(colnames(dat))
[1] "SS2_15_0048_A3" "SS2_15_0048_A6" "SS2_15_0048_A5" "SS2_15_0048_A4"
[5] "SS2_15_0048_A1" "SS2_15_0048_A2"
其中SS2_15都是一样的,Pxx也不需要管,重要的是中间的0048、0049,表示两个384孔板编号
那么如何提取?
使用strsplit函数,strsplit(x, split, fixed = FALSE) ,需要注意两点:
字符串切分后,返回的是一个列表,如果要再还原成字符串,需要用
unlist()默认情况下它是使用正则表达式的,如果不想用,可以指定
fixed = TRUE> unlist(strsplit("a.b.c", ".")) [1] "" "" "" "" "" > unlist(strsplit("a.b.c", ".", fixed = TRUE)) [1] "a" "b" "c"
# 方法一:纯base包(思路就是:将拆分得到的list变成数据框)
options(stringsAsFactors = F)
plate=do.call(rbind.data.frame,strsplit(colnames(dat),"_"))[,3]
# 方法二:stringr包
library(stringr)
plate=str_split(colnames(dat),'_',simplify = T)[,3]
最后新建细胞的属性信息
主要使用cutree剪下来的层次聚类信息、细胞板批次信息、每个样本的基因表达信息
前两个已经具备,下面进行第三个:每个样本的基因表达信息
# 还记得之前对基因进行过滤时,我们是对行进行操作
apply(a,1, function(x) sum(x>1) > floor(ncol(a)/50))
# 这里检测每个样本中有多少基因是表达的,count值以1为标准,rpkm值可以用0为标准
n_g = apply(a,2,function(x) sum(x>1))
# 对于单细胞转录组,一般会有超过半数的基因不会表达(这个在下面构建完数据框还可以再看一下)
可以构建数据框了:
meta=data.frame(g=group_list,plate=plate,n_g=n_g)
# 然后再添加一列,目前用不到,后续会介绍
meta$all='all'
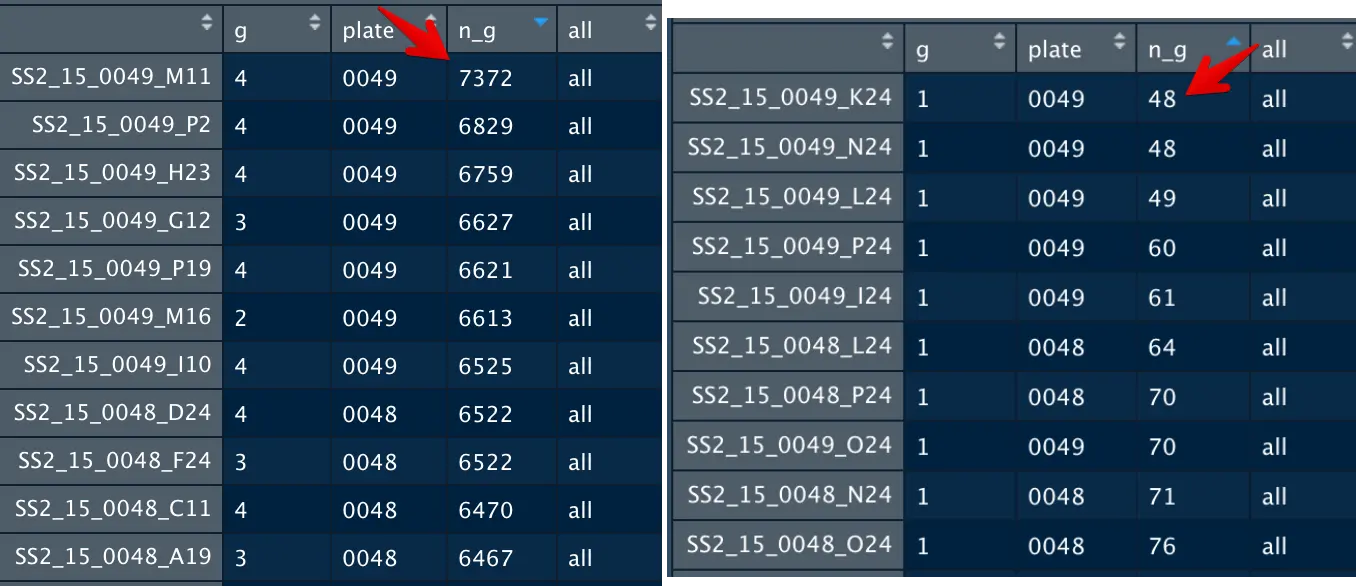
可以看到细胞中检测到表达的基因最多有7372个,最少才几十个,而我们总共有12000多个基因