145-一个复制粘贴引发的有趣小错误及思考
刘小泽写于19.11.5 这次的推送要感谢小郭(帮我看到了一个非常容易忽视的错误)和花花(帮我处理了一个简单但有用的脚本),又让我有了新的发现
问题是这样滴
我的需求
我想下载SRA 数据,然后需要构建一个配置文件(也就是一列是SRA ID),一列是样本名称。这样做的目的是为了后面使用fastq-dump 进行SRA转fq文件的结果更易懂
# 比如原来的一个sra是SRR391032.sra。如果只使用默认的fastq-dump参数,结果就是
SRR391032.1.fastq.gz
SRR391032.2.fastq.gz
# 但这样的数据多了,我们就分不清哪个数据对应哪个样本,于是需要在转换过程中就将样本名对应到fq文件上
# 例如构建这样一个config,第一列是SRR ID,第二列是sample
# config文件就长这样(举个例子)
SRR391032 WT-1
SRR391033 WT-2
SRR391034 TRT-1
SRR391035 TRT-2
现在找到GEO:https://www.ncbi.nlm.nih.gov//geo/query/acc.cgi?acc=GSE102339
进来就能看到一些sample和GSM的对应信息:

然后去获得SRR和GSM的对应信息:
https://www.ncbi.nlm.nih.gov/Traces/study/?acc=SRP114984
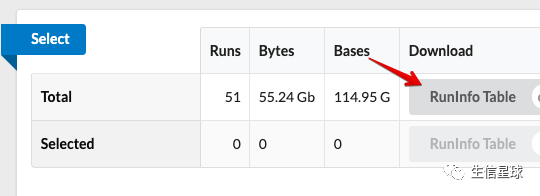
打开之后是这样(其中就包含了SRR和GSM的对应信息):
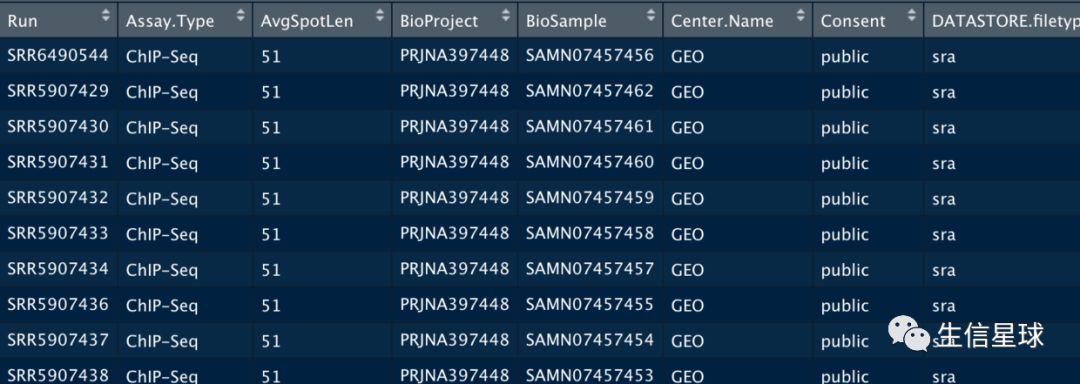
想法就是:将这两个文件通过共同的GSM编号对应起来
我的做法
step1:获得GSM与样本信息的对应=》GSM-to-sample.txt
像👇这样:从Samples信息的底部直接复制到顶部,然后粘贴到文本编辑器Sublime中,然后保存为GSM-to-sample.txt

然后读到R中
step2:获得GSM与SRR的对应
也是将之前下载的SraRunTable.txt读到R中
step3:准备找二者的对应关系
————————— 分界线 ——————————-
但就在这时,我在mac上查看了step1的这个文件:GSM-to-sample.txt
less -SN GSM-to-sample.txt
看到是52行,和网站上一致
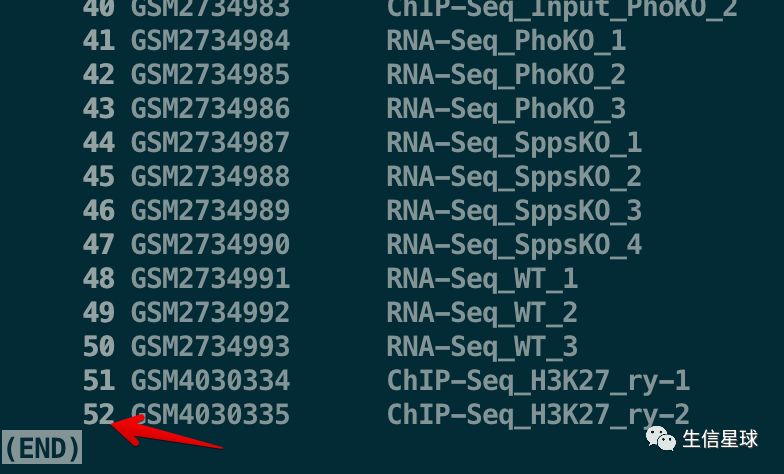
强迫症地想再次确认一下行数,就出了问题
cat GSM-to-sample.txt| wc -l
# 51
这两个竟然不一样,差了一行!很奇怪,想了很久,复制过来不应该出错,而且less和读到R中都是52行。另外我单独统计了第一列GSM号
cat GSM-to-sample.txt | cut -f1 | wc -l
# 52
小郭~复制粘贴少一行的解答
长期从事Linux工作、具有丰富debug经验、孜孜不断学习的小郭👏一眼看出了问题:
”是不是因为少了一个换行符“
真是一语点醒”梦中人“(其实我不困🤡)
对啊,想起来我复制过来的最后一行好像真的没有换行符,我重新vim编辑了一下这个文件,在最后一行敲了一下回车,然后再删掉最后的空行(这个看似没有任何改变的操作,其实给最后一行加了一个换行符)
然后再次检查:
cat GSM-to-sample.txt| wc -l
# 52
另外,如果是linux环境,还可以通过cat -A检查最后时候具有换行符,会看到:
cat -A GSM-to-sample.txt
”为什么less命令查看就是52行呢?而wc统计就会不识别“
这个就可能是不同的命令识别机制了,
wc -l统计的可能就是换行符(当然我也没有仔细去了解)
那么这个问题应该如何避免呢?
可以直接去到Linux环境,先输入一个cat >GSM-to-sample.txt
然后它会等待你的输入,将复制的结果直接粘贴进来,最后敲一下回车,再ctrl + C 退出,这时的文件也是完整的52行
总而言之,一定要确定最后一行有一个回车的操作,否则很有可能最后一行没有换行符,导致某些统计出错
花花~价值一元钱的脚本
现在有了两个完整的文件,就要进行SRR与样本信息的对应了
我当时在思考上面的复制粘贴问题,为了省事,就花钱”雇“了花花帮我写一个小脚本
脚本是这样的:
rm(list = ls())
x1 =read.csv("~/Downloads/SRA-to-GSM.txt")
> x1[1:4,1:4]
Run Assay.Type AvgSpotLen BioProject
1 SRR6490544 ChIP-Seq 51 PRJNA397448
2 SRR5907429 ChIP-Seq 51 PRJNA397448
3 SRR5907430 ChIP-Seq 51 PRJNA397448
4 SRR5907431 ChIP-Seq 51 PRJNA397448
x2 = read.csv("~/Downloads/GSM-to-sample.txt",sep = "\t",header = F)
> head(x2,3)
GEO_Accession V2
1 GSM2734944 ChIP-Seq_Ez_WT_1
2 GSM2734945 ChIP-Seq_Ez_WT_2
3 GSM2734946 ChIP-Seq_Ez_WT_3
colnames(x2)[1] ="GEO_Accession"
x3 = merge(x2,x1,by = "GEO_Accession")
# 关于merge函数,花花的解释是:”弱肉强食,谁放在前面谁是老大“,于是将x2放在前面就表示在x2的基础上添加x1的内容
> x3[1:4,1:4]
GEO_Accession V2 Run Assay.Type
1 GSM2734944 ChIP-Seq_Ez_WT_1 SRR5907429 ChIP-Seq
2 GSM2734945 ChIP-Seq_Ez_WT_2 SRR5907430 ChIP-Seq
3 GSM2734946 ChIP-Seq_Ez_WT_3 SRR5907431 ChIP-Seq
4 GSM2734947 ChIP-Seq_Pc_WT_1 SRR5907432 ChIP-Seq
config <- data.frame(srr=x3$Run,sample=x3$V2)
> head(config)
srr sample
1 SRR5907429 ChIP-Seq_Ez_WT_1
2 SRR5907430 ChIP-Seq_Ez_WT_2
3 SRR5907431 ChIP-Seq_Ez_WT_3
4 SRR5907432 ChIP-Seq_Pc_WT_1
5 SRR5907433 ChIP-Seq_Pc_WT_2
6 SRR5907434 ChIP-Seq_Ph_WT_1
没想到这里又出现了一个BUG!
之前的GSM和样本对应关系是x1(经上面的一顿操作,确实是52行);GSM和SRR的对应关系这里竟然只有51行(这个也没错,的确是51行)。那么就说明存在一个样本不存在SRR

到底是哪个在捣乱?找出来!
使用找差集的函数setdiff ,依然符合之前花花说的”弱肉强食“道理,谁放在前面谁是老大
这里的x2要多一行,所以它是老大
> setdiff(x2$GEO_Accession,x1$GEO_Accession)
[1] "GSM4030334"
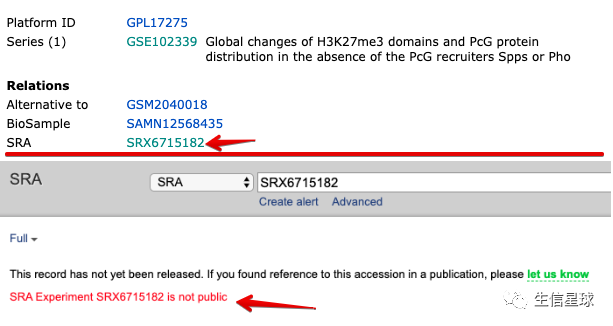
在GEO官网找到这个GSM4030334:看一下https://www.ncbi.nlm.nih.gov/geo/query/acc.cgi?acc=GSM4030334
结果发现这么一句话: