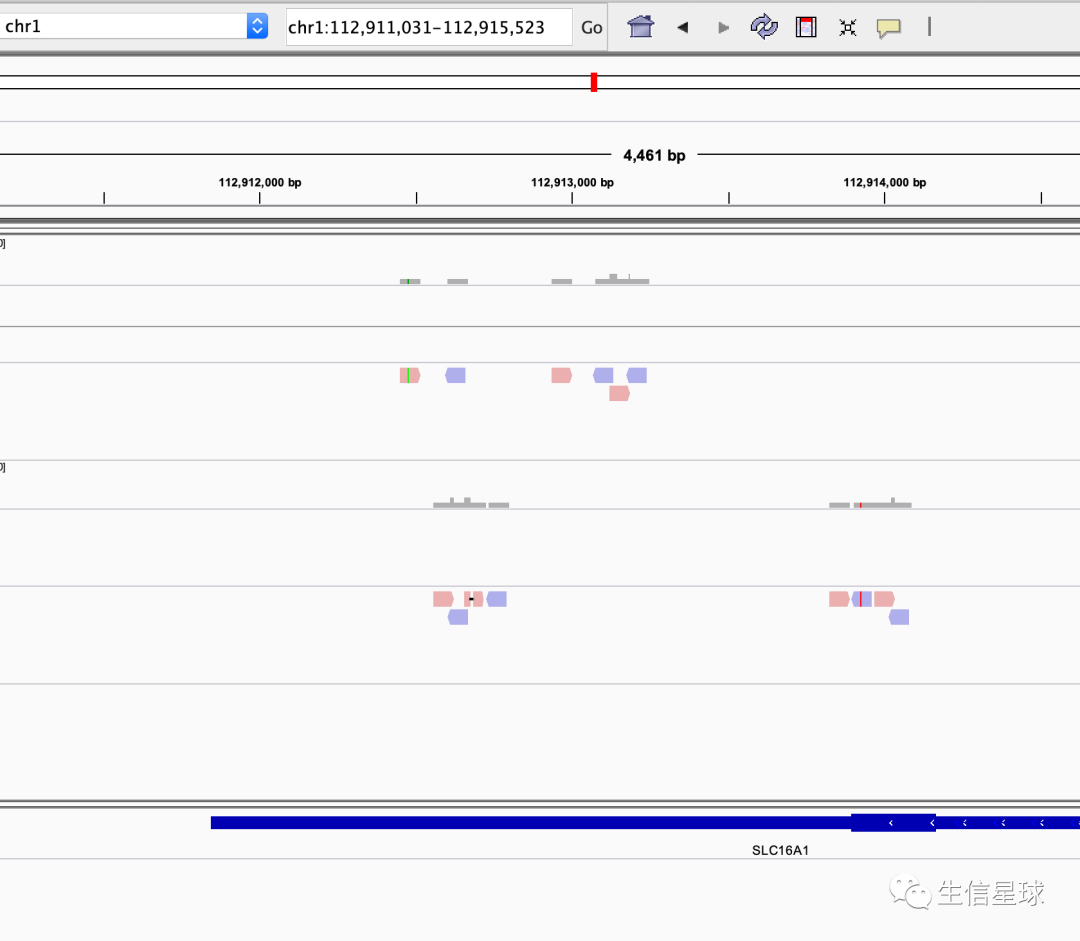
143-IGV加载很久很烦人?三步帮你解决!
刘小泽写于19.10.28
相信大家都遇到过,这个烦人的loading~
这是因为IGV的设计者默认全球都能访问他们的网站,但是,广大的国内用户做不到啊!
IGV启动每次都要加载一下基因组和注释文件,我们每次都要在loading上面浪费大量的时间,那么这个问题怎么破?
思前想后,我认为这个是一条最快的途径
第一步 断网
网络是loading的根源,断了网,它想访问也没用
第二步 重新打开IGV
一定会出现一个“报错“,说找不到这个基因组文件。但这个报错我们选择无视他,然后很快就能打开这个程序
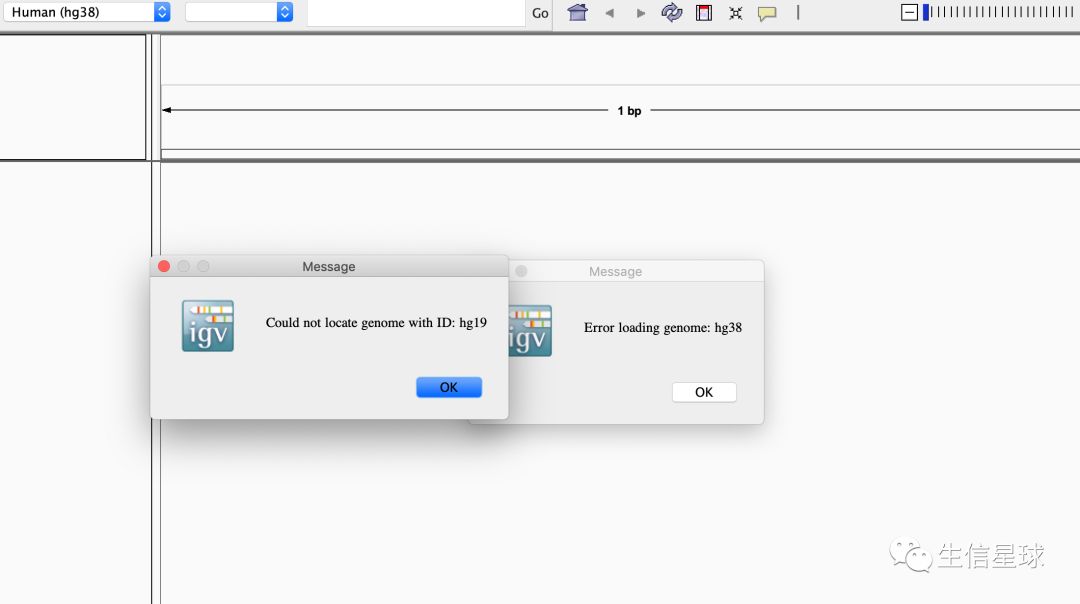
打开以后,就可以重新开启网络了
第三步 自己从本地加载基因组,一劳永逸
以人类基因组为例:
# 最好先用samtools 的faidx构建一个.fai索引文件
# for hg19 genome
cd ~/reference/genome/hg19
wget -c http://hgdownload.cse.ucsc.edu/goldenPath/hg19/bigZips/hg19.fa.gz
gunzip hg19.fa.gz && rm hg19.fa.gz
samtools faidx hg19.fa
# for hg38 genome
cd ~/reference/genome/hg38
wget -c http://hgdownload.cse.ucsc.edu/goldenPath/hg38/bigZips/hg38.fa.gz
gunzip hg38.fa.gz && rm hg38.fa.gz
samtools faidx hg38.fa
把fa和它的索引.fai放在本地,然后只需要通过Genomes=>Load Genome from File,导入FASTA文件
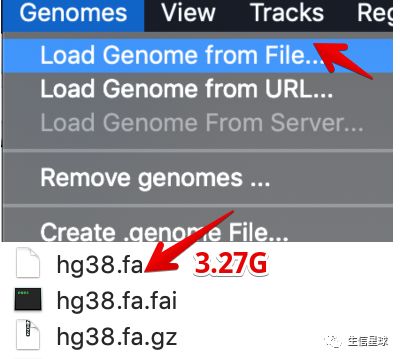
另外,还需要基因的注释,即加载基因组GTF文件
例如下载人类基因组基因注释文件:
wget ftp://ftp.ebi.ac.uk/pub/databases/gencode/Gencode_human/release_32/gencode.v32.annotation.gtf.gz
gunzip gencode.v32.annotation.gtf.gz
File => Load from file => 选择解压后的GTF文件,这是为了能看到基因的信息(如下图底部记录)
基因组和GTF都有了,就可以载入bam文件查看了
不过好像不能按照基因名查找,但是查看bam文件是绰绰有余了