025-转录组那些事儿
刘小泽写于18.8.19 之前写过三次关于转录组的实战小文,但是说实话,的确当时还不理解其中的含义,只是想跑个流程,更别说脚本优化了。就像刚买来一部心仪的电子产品,只想着拆开包装,嗅一下新机的味道,看看做工怎样,手感如何,但是它的内涵却不曾理解。后来学了其他的组学,接触久了,发现不深入了解,自己是无论如何都发掘不了它的潜力。
转录组火起来的原因主要是它能结合高通量测序,快速准确地识别转录本,进行表达定量,当然这也是它的核心功能。一般常见的转录组分析是找差异基因、协同变化基因、标记基因、融合基因、新转录本、可变剪切。结合R语言进行可视化、功能注释、网络分析。它既可以单枪匹马,也可以为别的组学打辅助。
好的结果离不开设计
差异来源?
- 样本差异:选取基因表达水平差异明显的不同组织细胞
- 处理差异:处理组和对照组相比,可能由于物理方法(物理损伤、照射等)、化学方法(药品刺激、抑制剂使用等)、生物方法(细菌、病毒侵染等)
- 剂量差异:做药物实验时,需要设计处理组各个药物剂量梯度,来验证药物的作用范围和效力
- 时间差异:时间不同,结果不同,找到特定时间点的影响结果或者分析某个发育现象与时间的关系
要测什么?
mRNA:最常见的转录组测序,建库一般选200-300bp的片段,150或125PE测序
microRNA:将microRNA分离出来直接单独测序
IncRNA:长链非编码RNA,有正向、反向转录,要进行链特异性建库
关于链特异性建库:作用就是测序过程保留转录本的方向信息,让我们知道转录本是来自正义链还是反义链。方便后来区分不同的IncRNA类型以及它的定位,可以更准确获得基因结构和表达信息
能干什么?
- 比较mRNA水平
- 同物种同组织不同处理:研究不同条件下基因的表达差异
- 同物种异组织:不同组织中基因的表达差异
- 同组织异物种:基因进化上的关系
- 以上三种可以加上时间变化:研究不同发育/用药时期基因表达差异
- 基因互作:大量样本建立基因的网络关系,找出通路,发现功能
- 表达模式:大量样本进行分类,发现与性状相关的基因,对样本进行预测
怎么提取?
原核生物大部分是核糖体RNA(rRNA),它的mRNA只占据了1-5%。要测它的mRNA,需要先提取纯化。
- 提取:大多数动植物组织样品,使用Trizol试剂即可;多糖含量丰富的植物,可以用多糖多酚试剂盒;脂肪组织可以用QIAGEN的RNeasy lipidmini kit ;
- 纯化:真核生物纯化mRNA,是利用它的3‘端polyA,采用oligoT磁珠将其富集纯化。但是原核没有polyA,因此需要先去除total RNA中的rRNA,需要使用去rRNA试剂盒(Ribo-Zero或KAPA试剂盒),另外对于要测物种IncRNA的实验,如果有适用的试剂盒就用,否则不适用就会影响下游数据质量
检测是否合格的指标:RNA总量、RIN值、OD260/280以及真核28S/18S、原核23S/16S。RIN值越高,28S/18S越接近2表示提取的RNA完整性越好。RIN值高于6.5可以做建库准备,太低影响准确度。有一些昆虫或者水生动物没有28S条带,因此RIN值不能作为参考,但是18S的前基线平稳即可
重复、深度、长度?
重复
- 生物学重复:不同的生物样本做同样的实验,想要发现组内差异
- 技术重复:一个生物样本测定多次
一般生物学重复要保持3以上,另外重复之间的Spearman相关系数要大于0.9(遗传背景不一致的相关系数要大于0.8)
另外,日常公司所说的“样本数量”=生物处理数*重复数,比如你有对照和处理组,各有三个重复,那么就是6个样本,当然测序分析的费用也是按样本收取
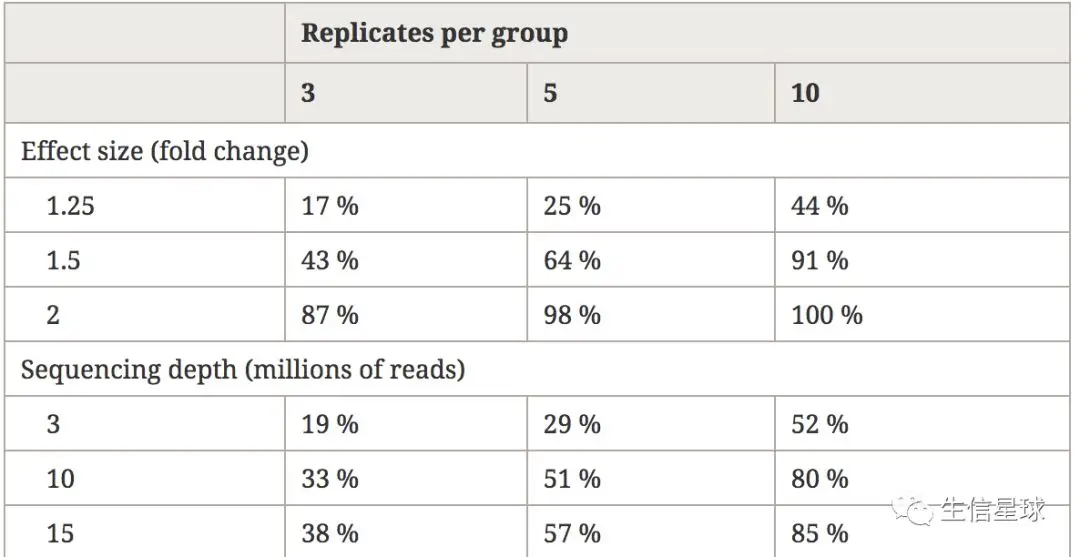
有一篇文章用酵母做过实验,doi: 10.1261/rna.053959.115,结果发现,随着重复的增加,找到差异基因越多;要筛到90%以上的差异表达基因,需要30个重复;其实实际分析,也不需要这么多的差异基因,使用合适的软件如edgeR或Deseq,可以控制假阳性率,即使样本重复数比较低,筛出的差异基因可信度还是比较高的。两个结论:生物学重复至少6个;对于每个实验处理要找到大部分(大概是80%以上)差异基因,至少12个生物学重复
根据文章https://doi.org/10.1186/s13059-016-0881-8当重复数为3时,差异倍数(Fold Change,FC)为1.5的基因只能找到43%。另外差异倍数较大的(FC>4)一般都能被覆盖到
深度
指的是测序得到的总碱基数与待测基因组大小的比值。深度越大,得到的reads条数越多,碱基越多。鉴定表达量中等深度即可,PE 150的reads数20M,测6G数据
结论就是:要为了找差异,花同样钱,多测样本好过加大深度
长度
多测一些长片段可以提高比对效率和转录本识别率,意思就是目标越大越易寻找,尤其对于基因组注释不好或者没有参考基因组的物种,双端测序加上长reads,会增加结果准确性
批次效应?
也许你见过它的分身"batch effect”。它是怎么回事呢?
不同测序平台的数据,同一个平台不同时间或者不同lane上产生的数据,同一样本不同时期,不同试剂做的同一样本等等,这些条件下产生的数据都是批次效应。简单说,就是你的数据量比较大时就容易出现。
是什么?
最常见的就是公司给你测的时候,不是放在一个测序仪上,对照组和实验组分开放置,比如对照是敏感组,实验组是抗性组,先测了抗性组,后来才测了敏感组,结果确实分析出了许多的差异基因。但差异基因是准的吗?会不会有可能是由于敏感组后来测的时候又发生了一些变化呢?说不准,但的确这里上机测序的时间成了干扰因素。要减少批次效应,一大方案就是选择支持更多样本的测序仪,例如NovaSeq一次建库就能容纳96*4个样本
尤其在分析公共数据库时,整合多个不同测序平台数据一起分析差异,这时很容易引入批次效应
如何检测?
怀疑哪个因素产生了干扰,就把它标记出来,比如怀疑时间产生了影响;然后对差异基因聚类分析,看看与时间前后是否相关,若相关就存在批次效应
如何校正?
详细内容可以看这本书https://genomicsclass.github.io/book/第10章
分析模式

基因组比对:有参考基因组,想分析新转录本
注释信息不是很完善,或者想找一些非编码RNA
一般步骤:测序reads比对到基因组=》基于比对结果组装转录本=〉基因/转录本表达定量=》差异或富集分析
转录组比对:有参考基因组,分析已知转录本
参考基因组注释较完善,如人、小鼠等模式生物,带着明确的目的去分析已知基因在样本中的表达。这种模式最简单、快捷
一般步骤:测序reads与转录本比对=》转录本定量=〉差异或富集分析
转录组组装后比对:没有参考基因组,或者有组装质量不好的,需要自己组装转录本(应用场景少,不适合入门)
一般步骤:测序reads进行De novo组装=》reads比对到组装的转录本=〉转录本表达定量=》差异或富集分析
好的结果离不开分析
一、数据预处理
原始数据:Illumina测序仪下机的数据通常为Bcl格式,然后公司使用Bcl2Fastq软件,根据Index序列分割转换成每个样品的Fastq文件,用户拿到的就是fastq格式的原始数据。
质控:使用fastqc,查看碱基质量、接头情况、GC含量、序列长度、重复序列等
过滤:一般需要去掉低质量碱基或者未识别碱基(N)太多的reads;另外如果测序文库的插入片段太短,比如insert size=50,但采用PE 150测序,read1和read2就会测到接头,所谓的“测通“就是这意思。此时需要去掉接头序列
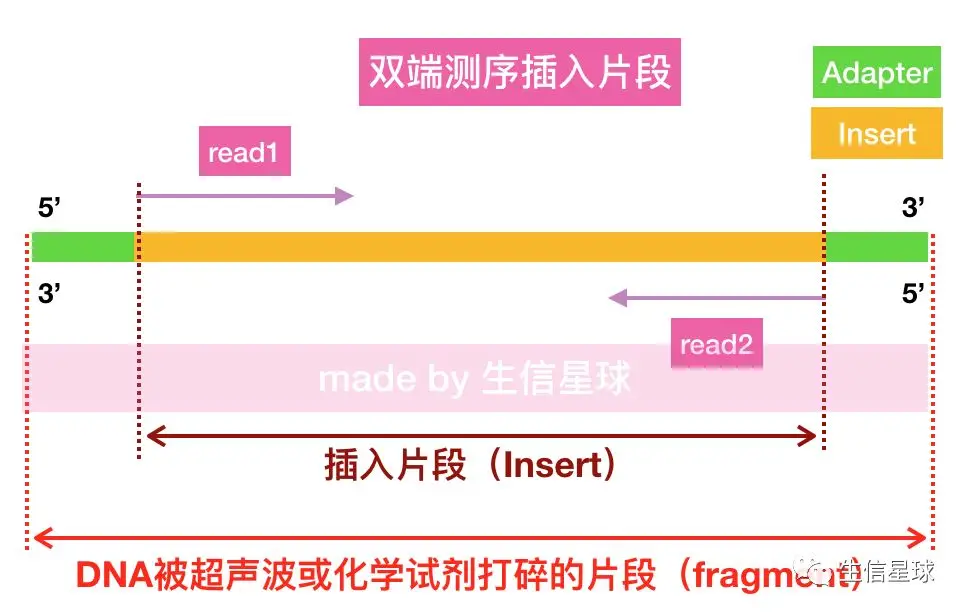
采用Trim Galore或者trimmomatic进行过滤;又或者采用国内的fastp软件,直接将质控和过滤整合,输出文件就是clean data
二、比对
虽然双端测序一次测了头和尾,但是并不能将整个mRNA转录本覆盖。我们结果得到了几百万条reads,要是知道哪条reads来自哪个转录本就能有的放矢,下面计算reads表达量也就知道了某个转录本或者基因的表达量。 将测序reads与参考转录本/基因的比较匹配过程就是比对,可以说mapping 或者alignment
由于DNA转录得到mRNA时将内含子切除,因此mRNA反转录得到的cDNA不一定十分完美的还原回原基因组,相当大一部分会被分开。因此原来为基因组比对设计的软件如bwa可能效果会下降,可以采用专门为转录组开发的比对软件Hisat2、STAR,可以找到剪切位点。当然,如果只为了寻找差异基因,可以用bowtie2以及更快的非比对软件salmon、sailfish、jellyfish。
文章A survey of best practices for RNA-seq data analysis中做了几款主流的比对软件Tophat、Hisat2、STAR评测,结果发现:
- Hisat2比对回去的拼接点比较少,但是找的拼接点成功率高(也就是说,它比对的量少但质优)。
- STAR比对到基因组上唯一的reads数最多,对于双端reads,比对不上的STAR就移除,不会选择妥协只比对单端;它的稳定性最好,体现在处理较长reads和较短reads的结果不会波动太大;STAR容忍性比较好,容易接受错配碱基和soft-clipping(没比对上的不去除,只标记出来),只为帮更多reads“找到家”
- 速度方面,Hisat2比STAR快2.5倍,比上一任tophat快约100倍
看似简单的比对过程,就是帮150bp的reads找到家,其中可能还要让reads付出点“被分割”的代价。但是, 基因组有多大?人类的是3G,也就是30亿碱基,一个150bp对于整个基因组来说,简直不值一提,要从头一个一个比对吗?姑且这样可以,那么我们有多少reads?一般6G数据,150PE,会有20Mreads(=60亿/150/2),也就是2000万条reads。这该怎么办?怎样保证高效和低错误率?
reads是测序仪决定的,是固定的,就是这么多,就是这么长。那么我们只能从参考基因组入手,怎么让他找的快?这里就用到了一个算法——BWT(Burrows–Wheeler_transform),他其实是一个压缩技术,将原来文本转换成一个相似的文本,转换后让相同的字符位置连续【https://blog.csdn.net/luanzheng_365/article/details/78575429】 。使用这个算法,将基因组变成了一个索引index,而我们要查找的序列就是索引中的一个子集,这样比对的任务就不再是将reads从头到尾和基因组去比较,而是转换成了把子集reads和索引index去比较,做到了有的放矢。
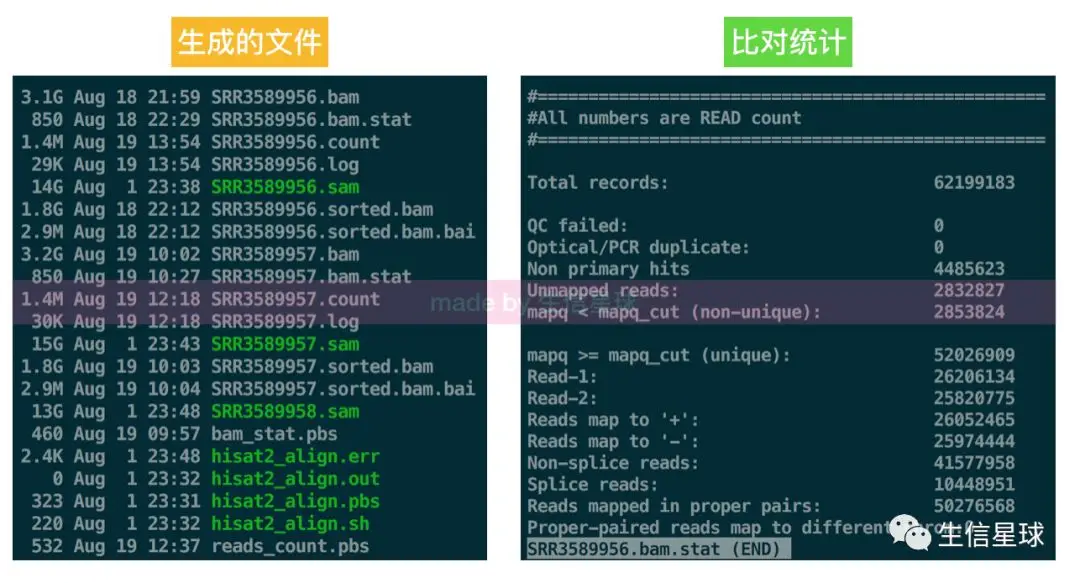
比对完我们需要的是bam文件,然后使用bam还可以做其他一些比对统计,或者导入IGV查看
三、定量(三个水平)
基因水平
常用htseq-count、featureCounts、bedtools(multicov)、Qualimap、GenomicRanges,它们根据read和基因位置的重叠区域判断read的归属。
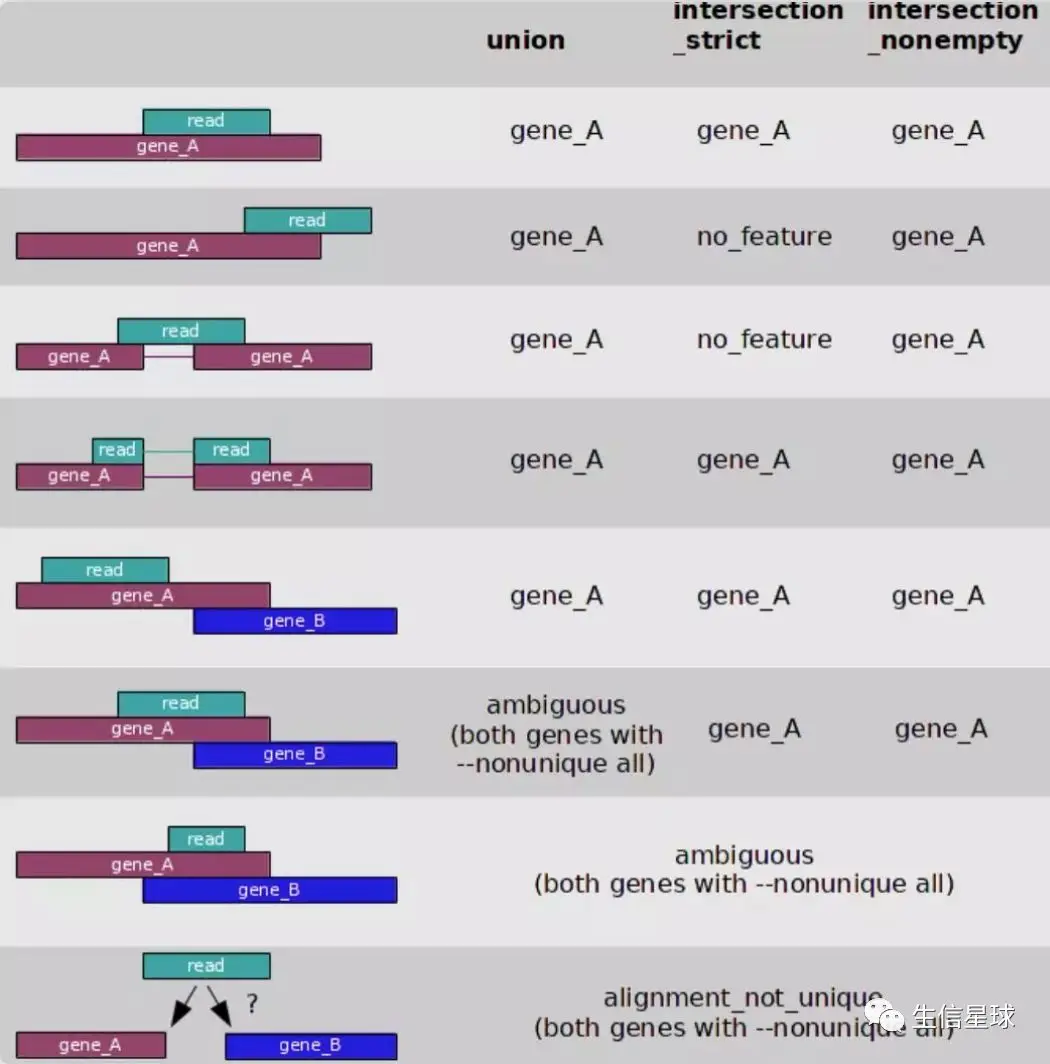
以
htseq-count为例,它默认采用union方式进行统计哪些reads分配到哪个基因上。从图上看,软件对前几种都容易判断,但是后三种出现了多比对现象(multi-mapping reads),这时各个定量软件就出现了差别,htseq-count选择无视这种情况,但是Qualimap选择将geneA、B都算上。这个软件性能不错,但就是速度慢。如果有许多样本等待处理,那么
featureCounts或许是不错的选择。featureCounts被整合到了subread中,它对于多重比对的reads并不像htseq一样全部丢弃,而是根据比对的不同区域大小比较,最终选择排除、全部或部分计算每个样品进行计数后,都是一个个分散的文件,需要将他们合并成一个表达矩阵,行为基因名,列为样品名,中间是计数结果。对于这个矩阵matrix,后期分析需要再标准化【一般产生偏差的因素主要是:基因长度、测序深度、GC含量、测序仪系统误差】。标准化的方式有:RPKM(单端测序用的多)、FPKM(目前主流)、TMM、TPM。也有的软件会自动进行标准化。
另外,有的软件需要标准化后的矩阵,有的不需要(如DESeq2)
外显子水平
在基因水平之上,又分析的差异的外显子,使用DEXSeq的dexseq_prepare_annotation.py脚本。另外需要提供无重叠的外显子区域gtf文件
转录本水平
基于比对的一般采用Stringtie、eXpress;不比对直接得计数结果的kallisto、sailfish、salmon
第二部分写于18.8.21,使用文章数据在linux上跑出来表达矩阵 第三部分准备用R语言进行下游的特别分析,包括可视化、差异基因筛选、富集分析等
阅读文献
选取的文献是哈佛大学Lee L. Rubin团队于2015年发表在Cell Stem Cell中的Genome-wide RNA-Seq of Human Motor Neurons Implicates Selective ER Stress Activation in Spinal Muscular Atrophy,doi是:http://dx.doi.org/10.1016/j.stem.2015.08.003
文章梗概
运动神经元存活蛋白(Survival Motor Neuron, SMN)是人体内的泛表达蛋白,它的会导致脊髓性肌萎速症(Spinal muscular atrophy, SMA),SMA是由SMN1基因突变引起的,但是这个基因是广泛表达的基因,为什么运动神经元(Motor Neurons, MNs)偏偏是最易受影响的细胞类型之一?实验通过对照组和SMA患者诱导多能干细胞(IPSC)从而产生纯化的运动神经元,通过固定、抗体标记,然后进行了RNA测序研究。在患者运动神经元中发现了SMA特异性的变化,其中包括内质网(ER)应激通路的过度激活。功能研究表明,抑制内质网的应激反应能提高运动神经元的存活率。在患病小鼠中,使用ER应激抑制剂穿过血脑屏障可以保护脊髓运动神经元。因此,选择性激活内质网应激反应,导致了SMA患病个体的运动神经元死亡。
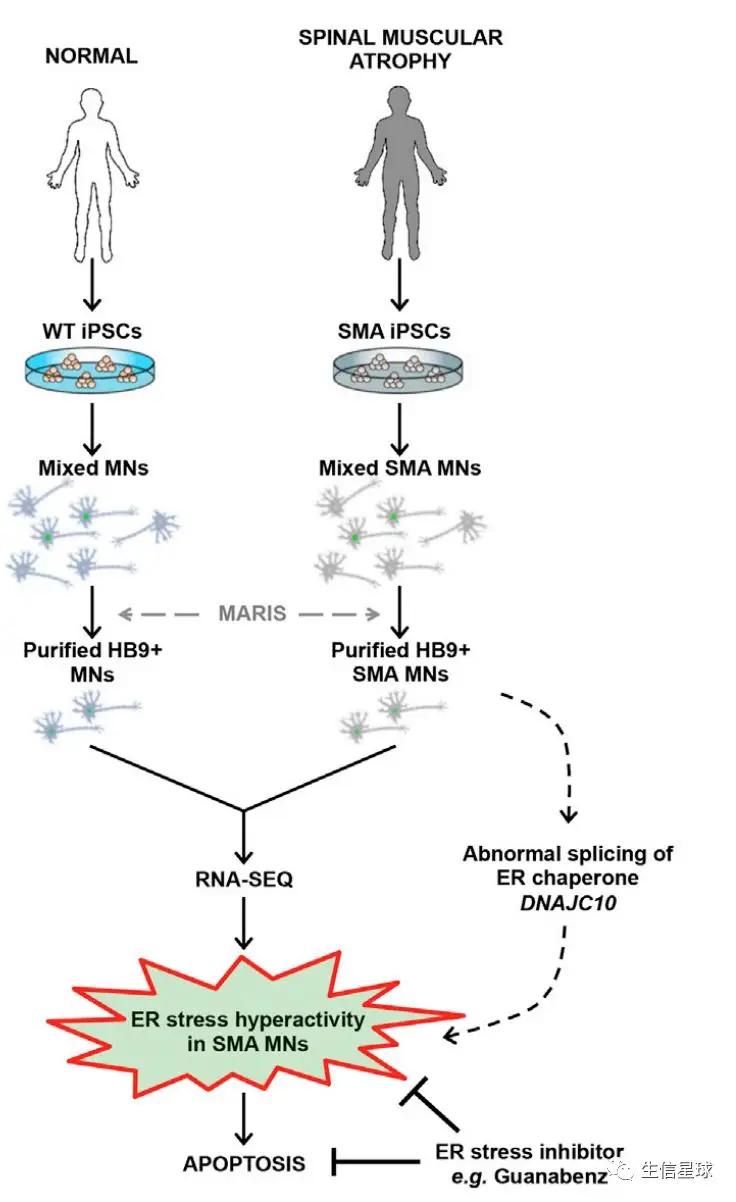
RNA-seq数据存储在GSE69175中

开始实战
万事之源,软件为先
#配置conda
conda create -n rna-seq python=2 samtools fastp sra-tools hisat2 rseqc -y
conda install -c bcbio htseq -y
conda install numpy pysam -y
CONBIN=/home/biosoft/miniconda3/envs/rna-seq/bin
配置好工作路径
RNA=/home/project/rna-seq
mkdir -p $RNA/{raw_data,clean_data,ref/{genome,gtf,index},align,stats,matrix}
RAW=$RNA/raw_data
CLEAN=$RNA/clean-data
GENOME=$RNA/ref/genome
GTF=$RNA/ref/gtf
INDEX=$RNA/ref/index
ALIGN=$RNA/aign
MATRIX=$RNA/matrix
STATS=$RNA/stats
下载测试数据
GEO数据库中的编号是:GSE69175
关于数据:
NGS测序数据一般会上传到SRA数据库里面,但是怎么从GEO数据库中找到SRA原始数据的下载地址?【GEO数据库地址:https://www.ncbi.nlm.nih.gov/geo/】

具体的层级关系是:SRP(项目)—>SRS(样本)—>SRX(数据产生)—>SRR(数据本身)
SRR数据库地址:https://ftp-private.ncbi.nlm.nih.gov/sra/sra-instant/reads/ByRun/sra/SRR/
##################################################################
################# Part 1: Data download #########################
##################################################################
# SRR2038215-SRR2038216: 对照
# SRR2038217-SRR2038218: SMN
cd $RAW
for i in `seq 15 18`;do
ascp -v -i ~/.aspera/connect/etc/asperaweb_id_dsa.openssh \
-k 1 -T -l200m anonftp@ftp-private.ncbi.nlm.nih.gov:/sra/sra-instant/reads/ByRun/sra/SRR/SRR358/SRR35899${i}/SRR35899${i}.sra ./ #使用ascp下载,嗖嗖嗖~
done
fastq-dump --gzip --split-3 *.sra #sra转为fastq
#以下3行原版来自生信技能树Jimmy
find $RAW -name "*.gz" | grep 1.fastq.gz >1
find $RAW -name "*.gz" | grep 2.fastq.gz >2
paste 1 2 > raw_conf #将read1和read2各自的合集再整合到一起,形成一个数据配置文件
cp raw_conf $CLEAN
质控过滤
这里使用的是fastp,同时融合了质控、过滤的模式; 同时也可以使用fastqc + trimmomatic/ trim_galore进行
##################################################################
################## Part 2: QC & trim ############################
##################################################################
source activate rna-seq
cd $CLEAN
#下面四行原版来自生信技能树Jimmy
cat raw_conf | while read id;do
line=(${id})
fq1=${line[0]}
fq2=${line[1]}
fastp -i $fq1 -I $fq2 -o out.$(basename $fq1) -O out.$(basename $fq2)-w 16
done
source deactivate
下载参考基因组和注释文件
人类参考基因组选择UCSC数据库;注释文件选择GENCODE,https://www.gencodegenes.org/
下载好基因组,需要构建基因组索引
如果自己的项目做非模式物种,可以用二代三代测序数据组装成参考转录组,例如trinity组装。如果做的物种有参考基因组,就方便一些,可以直接从相关数据库中下载参考基因组
##################################################################
############### Part 3: prepare ref & index ######################
##################################################################
##Download hg19
cd $GENOME
for i in $(seq 1 22) X Y M;do
wget http://hgdownload.cse.ucsc.edu/goldenPath/hg19/chromosomes/chr${i}.fa.gz;
done
gunzip *.gz
for i in $(seq 1 22) X Y M;do
cat chr${i}.fa >> hg19.fa
done
rm chr*
##或者也可以直接下载成品
#wget http://10.10.0.8/hgdownload.soe.ucsc.edu/goldenPath/hg19/bigZips/hg19.fa.gz
##Download GRCh38 https://www.gencodegenes.org/releases/current.html
cd $GTF
wget ftp://ftp.ebi.ac.uk/pub/databases/gencode/Gencode_human/release_28/GRCh37_mapping/gencode.v28lift37.annotation.gtf.gz
gunzip *.gz
## hisat2 index
cd $INDEX
hisat2-build -p 20 $GENOME/hg19.fa hg19 #-p为线程数
比对
##################################################################
######## Part 4: align & sort & index human samples:56-58 ########
##################################################################
source activate rna-seq
cd $ALIGN
for i in `seq 15 18`;do
hisat2 -t -p 20 -x $INDEX/hg19 \
-1 $CLEAN/out.SRR20382${i}_1.fastq.gz \
-2 $CLEAN/out.SRR20382${i}_2.fastq.gz \
-S SRR20382${i}.sam
samtools view -Sb SRR20382${i}.sam > SRR20382${i}.bam
samtools sort -@ 20 -o SRR20382${i}.sorted.bam SRR20382${i}.bam
samtools index SRR20382${i}.sorted.bam
rm *.sam
done
source deactivate
#关于排序:默认按染色体位置排序;-n根据read名排序;-t根据tag排序
使用了samtools的三件套:转换(view)、排序(sort)、建索引(index) 转换: -S指输入文件格式(不加-S默认输入是bam),-b指定输出文件(默认输出sam)【如果要bam转sam,-h设置输出sam时带上头注释信息】 排序: 对bam排序,-@ 线程数, -o输出文件 索引: 结果会产生.bai文件【必须排序后才能建索引~就像体育课必须从高到矮排好以后再报数】
基本信息统计
##################################################################
################ Part 5: basic statistics #######################
##################################################################
source activate rna-seq
cd $STATS
for i in `seq 15 18`;do
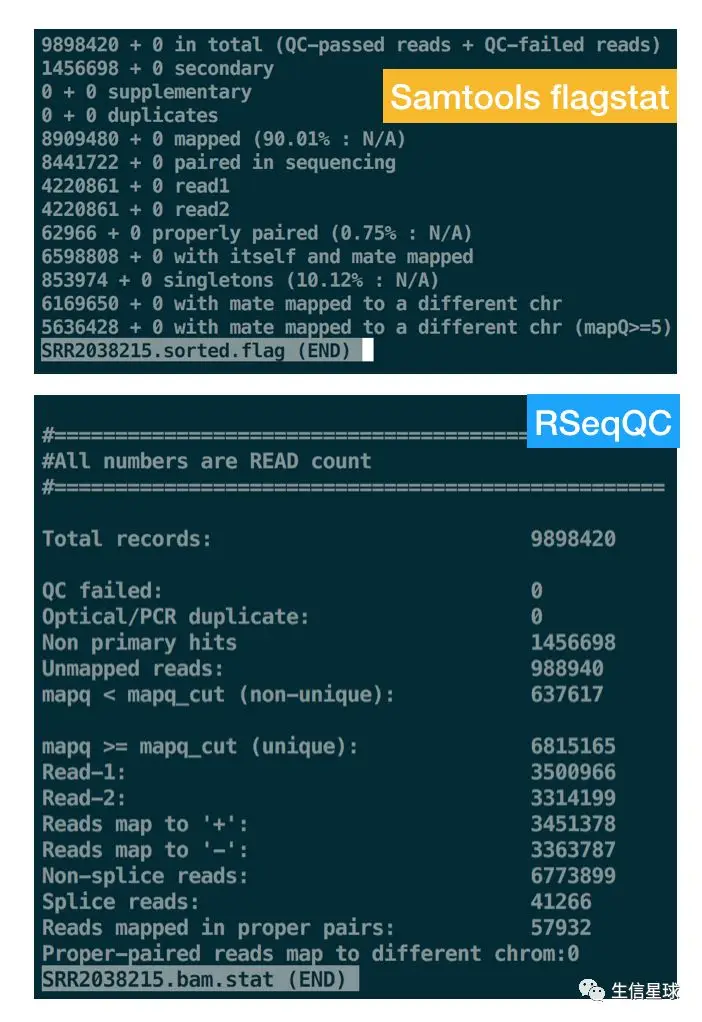
samtools flagstat $ALIGN/SRR20382${i}.sorted.bam > SRR20382${i}.sorted.flag
done
#如果想根据flag提取特定区域,比如想查看1号染色体100-10000的区域的信息
#samtools view -b -f 0x10 $ALIGN/SRR20382${i}.sorted.bam chr1:100-10000 > ${i}.flag.bam
#samtools flagstat ${i}.flag.bam
#使用RSeqQC统计
#先用bam_stat.py对bam文件统计,看下比对率
#bam_stat.py -i $ALIGN/SRR20382${i}.sorted.bam > SRR20382${i}.bam.stat
source deactivate
reads计数
基于基因组水平,可以用Htseq-count和featureCounts
Htseq-count:它是用python写的一个脚本,
conda install -c bcbio htseq -y安装好以后可以直接拿来用需要比对文件sam/bam格式;需要基因组注释文件gff/gtf格式。这两个文件的染色体名称必须相同,因为需要将比对位置和特征信息相匹配就要借助染色体名。 它的工作原理是:先通过bam文件找到reads比对上的外显子,然后去gtf文件中将外显子的基因ID进行统计,得到的结果就是未经标准化的表达量
source activate rna-seq cd $MATRIX for i in `seq 15 18`;do $CONBIN/htseq-count -s no -r name -f bam $ALIGN/SRR20382${i}.sorted.bam \ $GTF/gencode.v28lift37.annotation.gtf \ >SRR20382${i}.count 2>SRR20382${i}.log done source deactivatehtseq-count的
-s参数是strand意思,默认使用链特异性建库方式,也就是计算过程中要求read1只能和双端测序的其中一条比较,read2只能和另一条比较;我们一般设置no即可,表示每条read都和正负链比较一次;-r参数两个选择name和pos,数据根据位置或者read名称排序,默认name;-f参数输入文件格式bam/sam;-m参数一般也就是默认union模式结果每个样本输出一个count文件,其中包含了基因名和reads计数;另外,如果你看到文件倒数几行,会发现还有几行带文字的
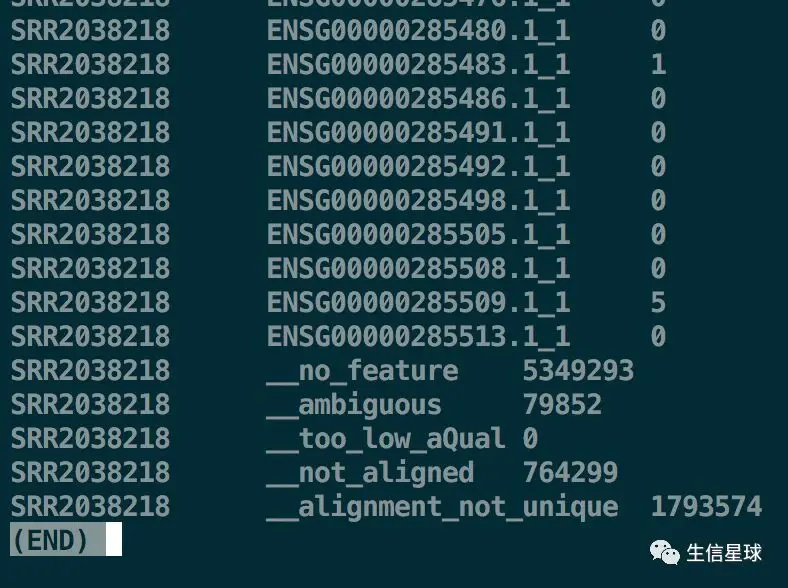
no_feature:比对区域与任何基因都没有重叠 ambiguous:比对区域与多个基因都发生重叠 too_low_aQual:比对质量低于设定阈值(默认是10) not_aligned:无法比对上 alignment_not_unique:比对位置不唯一
featureCounts:【相比于htseq更快】
source activate rna-seq cd $MATRIX begin=$(date +%s) for i in `seq 15 18`;do featureCounts -T 20 -p -t exon -g gene_id -a $GTF/gencode.v28lift37.annotation.gtf -o SRR20382${i}.feature.count $ALIGN/SRR20382${i}.bam done tim=$(echo "$(date +%s)-$begin" | bc) printf "time of featureCounts for 4 samples: %.4f seconds" $tim source deactivate #运行结果显示: #time of featureCounts for 4 samples: 366.5310 seconds-T参数设置线程,-p参数表示针对双端测序数据,-t 参数默认将外显子认定为基因组的一个feature(搜索特征),-g参数默认按照基因名来计数,-a参数输入注释文件,-o参数输出文件结果有两个,一个count文件,一个summary文件
对两个软件的结果进行合并
##合并htdeq生成的count文件到matrix.count cd $MATRIX/htseq perl -lne 'if ($ARGV=~/(.*).count/){print "$1\t$_"}' *.count | grep -v "_" >matrix.count ##合并featureCounts生成的count文件到matrix_2.count cd $MATRIX/featureCounts for i in `seq 15 18`;do cut -f 1,7 SRR20382${i}.feature.count | grep -v "^#" > SRR20382${i}.matrix sed -i '1d' SRR20382${i}.matrix done perl -lne 'if ($ARGV=~/(.*).matrix/){print "$1\t$_"}' *.matrix >matrix_2.count统计一下两个软件的计数之和
#统计featureCounts perl -alne '$sum += $F[2]; END {print $sum}' matrix.count #结果是5882943 #统计htseq-count,结果是786338
疑问🤔️:为什么这两个软件出来的结果相差这么大?
按说统计是没有问题的,我查看了以下二者表达量非0的基因数
#对于htseq-count
less matrix.count | grep -v "0$" | wc -l #结果为2811
#对于 featureCounts结果为72531